一、氨基,羧基和氨基酸
氨基:-NH2 氨基是基本碱基,大多数含有氨基的有机物都有一定碱的特性。氨基是一个活性大、易被氧化的基团。
羧基:-COOH。分子中具有羧(念suo)基的化合物称为羧酸。
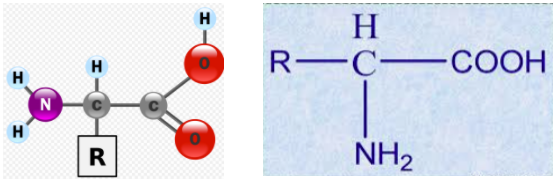
氨基酸,结构中含有胺基和羧基,并且胺基和羧基都是直接连接在一个-CH-结构上的有机化合,化学式是RCHNH2COOH。羧酸碳原子上的氢原子被氨基取代后形成的化合物。氨基酸分子中含有氨基和羧基两种官能团。下图是α-氨基酸的分子结构。经蛋白质水解后得到的氨基酸都是α-氨基酸,而且仅有二十二种,包括甘氨酸、丙氨酸、缬氨酸、亮氨酸、异亮氨酸、甲硫氨酸(蛋氨酸)、脯氨酸、色氨酸、丝氨酸、酪氨酸、半胱氨酸、苯丙氨酸、天冬酰胺、谷氨酰胺、苏氨酸、天冬氨酸、谷氨酸、赖氨酸、精氨酸、组氨酸、硒半胱氨酸 [和吡咯赖氨酸 ,它们是构成蛋白质的基本单位。
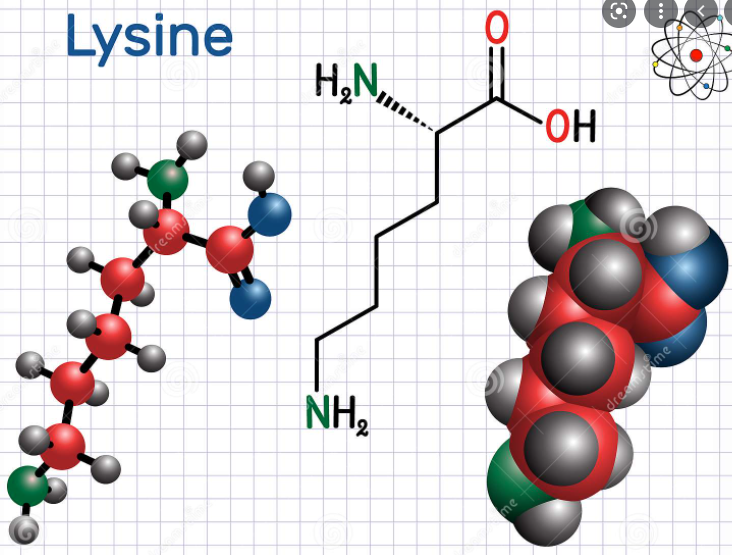
例如,赖氨酸,如下图
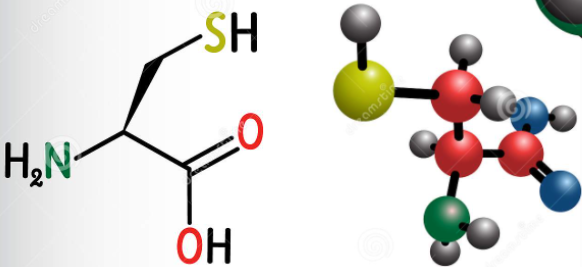
例如,半胱氨酸,如下图
二、肽键和多肽
肽键是一个氨基酸的羧基与另一个氨基酸的氨基缩水形成的共价键。肽键的形成过程如下图
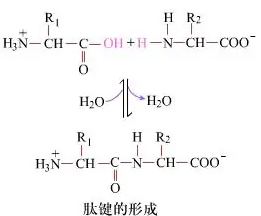
我们把由于去水形成的C原子和N原子之间的键就叫做肽键,也称为酰胺键,写作—CO—NH一,如下图中的红线所示
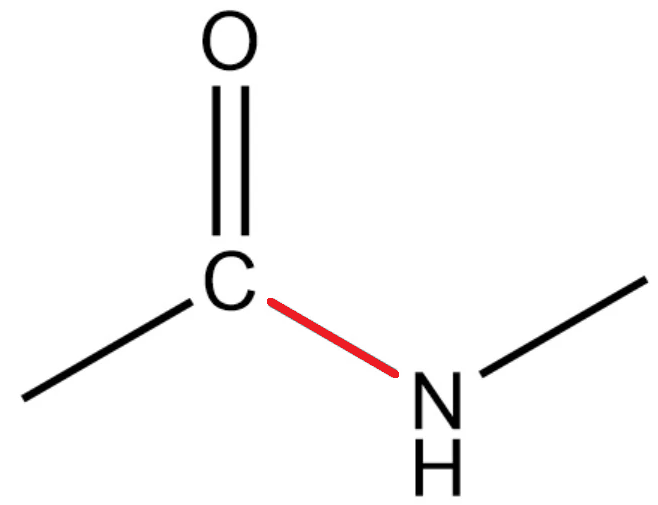
肽键中的原子由于可产生共振而表现出较高的稳定性。在肽键中C-N单键具有约40%双键性质。
最简单的肽由两个氨基酸组成,称为二肽。含有三个氨基酸的肽称为三肽,以此类推。肽链中的氨基酸由于形成肽键时脱水,已不是完整的氨基酸,所以称为残基。
多肽示例:

在蛋白质分子中,氨基酸之间以肽键连接起来,形成肽链。

三、蛋白质结构
蛋白质结构是指蛋白质分子的空间结构。作为一类重要的生物大分子,蛋白质主要由碳、氢、氧、氮、硫等化学元素组成。所有蛋白质都是由20种不同的L型α氨基酸连接形成的多聚体,在形成蛋白质后,这些氨基酸又被称为残基。蛋白质和多肽之间的界限并不是很清晰,有人基于发挥功能性作用的结构域所需的残基数认为,若残基数少于40,就称之为多肽或肽。一定数量的残基对于发挥某一生物化学功能是必要的;40-50个残基通常是一个功能性结构域大小的下限。蛋白质大小的范围可以从这样一个下限一直到数千个残基。
要发挥生物学功能,蛋白质需要正确折叠为一个特定构型,主要是通过大量的非共价相互作用(如氢键、离子键、范德华力和疏水作用)来实现;此外,在一些蛋白质(特别是分泌性蛋白质)折叠中,二硫键也起到关键作用。为了从分子水平上了解蛋白质的作用机制,常常需要测定蛋白质的三维结构。由研究蛋白质结构而发展起来了结构生物学,采用了包括X射线晶体学、核磁共振以及冷冻电镜(cryo-EM)等技术来解析蛋白质结构。
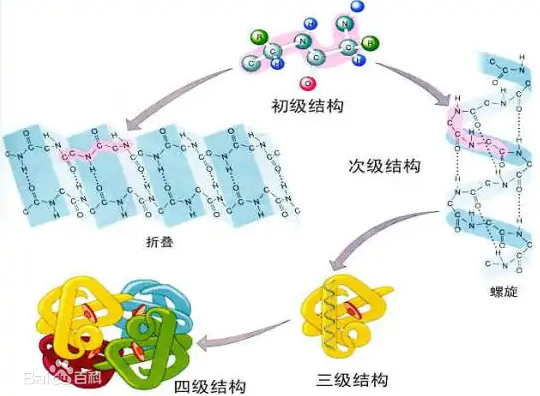
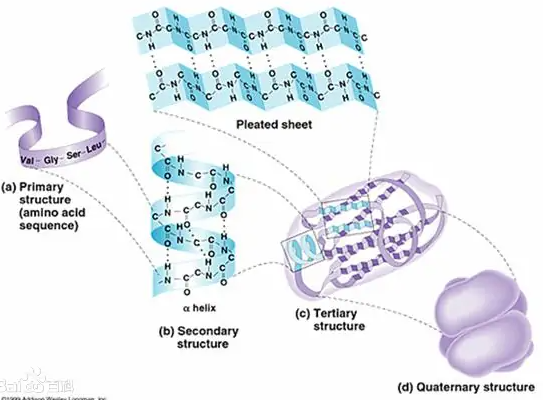
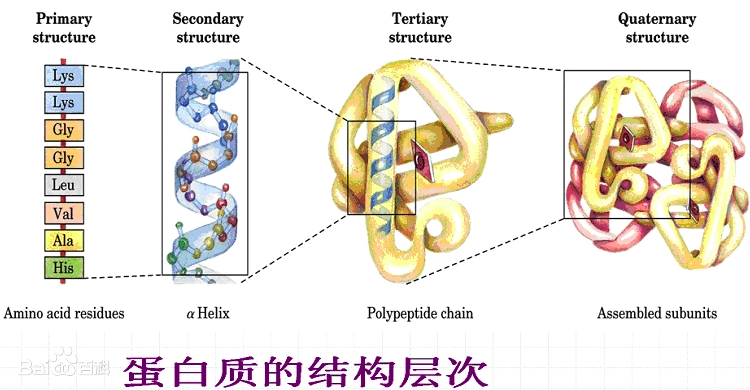
一级结构
肽或蛋白质的氨基酸序列(或残基序列)被称为蛋白质一级结构。蛋白质一级结构(primary structure)也包括二硫键的位置。

二级结构
蛋白质二级结构(secondary structure of protein)是指多肽主链骨架原子沿一定的轴盘旋或折叠而形成的特定的构象,即肽链主链骨架原子的空间位置排布,不涉及氨基酸残基侧链。蛋白质二级结构的主要形式包括α-螺旋、β-折叠、β-转角和无规卷曲(环)。由于蛋白质的分子量较大,因此,一个蛋白质分子的不同肽段可含有不同形式的二级结构。维持二级结构的主要作用力为氢键。一种蛋白质的二级结构并非单纯的α螺旋或β折叠结构,而是这些不同类型构象的组合。

三级结构
蛋白质三级结构(protein tertiary structure):蛋白质分子处于它的天然折叠状态的三维构象。三级结构是在二级结构的基础上进一步盘绕,折叠形成的。三级结构主要是靠氨基酸侧链之间的疏水相互作用,氢键,范德华力和静电作用维持的。(在二级结构的基础上借助各种次级键卷曲折叠成特定的球状分子结构的构象)。
四级结构
在体内有许多蛋白质含有2条或2条以上多肽链,才能全面地执行功能。每一条多肽链都有其完整的三级结构,称为亚基(subunit),亚基与亚基之间呈特定的三维空间分布,并以非共价键相链接,这种蛋白质分子中各亚基的空间排布及亚基接触部位的布局和相互作用,即寡聚蛋白中各亚基的空间排布,称为蛋白质的四级结构(Quaternary structure)。蛋白质亚基之间主要通过疏水作用、氢键、离子键等作用力形成四级结构,其中最主要的是疏水作用。
蛋白质折叠
折叠类型则指的是整体的结构排列类型,如螺旋束、β桶、和罗斯曼折叠,或是由蛋白质数据库结构分类中提供的不同的折叠。从一级结构到更高级结构的过程就被称为蛋白质折叠。一个序列特定的多肽链(折叠之前的蛋白质一般都被称为多肽链)一般折叠为一种特定构象(又称为天然构象);但有时可以折叠为一种以上的构象,且这些不同构象具有不同的生物学活性。在真核细胞内,许多蛋白质的正确折叠需要分子伴侣的帮助。
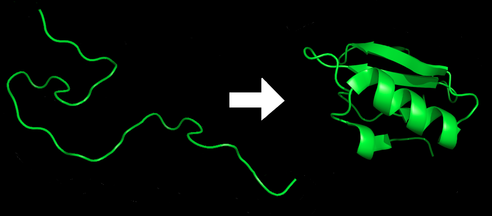
结构分类
对蛋白质结构进行分类的方法有多种,有多个结构数据库(包括SCOP、CATH、和FSSP)数据库提供不同的结构分类。分别采用不同的方法进行结构分类。存放蛋白质结构的PDB数据库中就引用了SCOP的分类。对于大多数已分类的蛋白质结构来说,SCOP、CATH和FSSP的分类是相同的,但在一些结构中还有所区别。
结构预测
测定蛋白质序列比测定蛋白质结构容易得多,而蛋白质结构可以给出比序列多得多的关于其功能机制的信息。因此,许多方法被用于从序列预测结构。
- 同源建模:需要有同源的蛋白三级结构为基础进行预测。
- “从头开始”(Ab initio):只需要蛋白质序列即可进行结构预测。由于运算量大,需要有超级计算机来进行,或采用分布式计算。
- 四级结构预测:主要是预测蛋白质-蛋白质之间的相互作用方式。
参考资料
[1] 百度百科
[2] 肽键的性质和肽的功能
[3] 维基百科:蛋白质结构