rMATs是一款分析RNA可变剪切事件的软件,其最大特点是考虑了replicate,还针对配对样本(在医学方面很多都是tumor和normal配对)建立了统计模型,如此以来,得到的结果相对来说也更好。具体可参考文献《rMATS: Robust and flexible detection of differential
alternative splicing from replicate RNA-Seq data 》,软件的下载和使用参见http://rnaseq-mats.sourceforge.net/index.html
在分析的到最终结果时,虽然软件官网有对结果的介绍,但看到结果开始一脸懵逼。

现结合自己的理解,记录下,防止以后又忘了
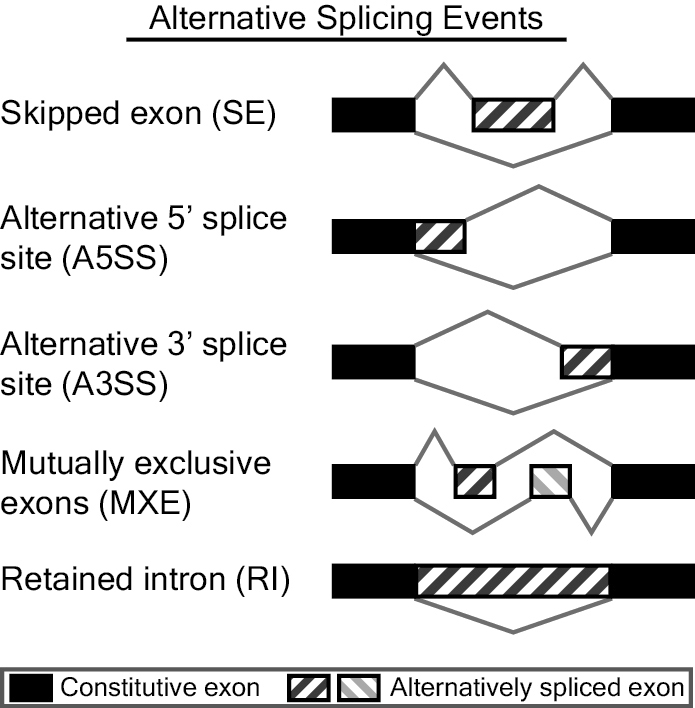
fromGTF.XX.txt系列:
应该是直接从GTF文件和数据文件读出的结果
SE:skipped exon
RI:retained intron
MXE:mutually exclusive exons
A5SS:alternative 5' splice site
A3SS:alternative 3' splice site
fromGTF.novelEvents.XX.txt:从数据文件发现的新的可变剪切事件
XX.MATS.JC.txt和XX.MATS.JCEC.txt是JC.raw.input.XX.txt和JCEC.raw.input.XX.txt经过统计模型分析后的结果,多了p值和FDR值
至于xx.txt文件头文件的含义,在biostar上已经有人回答的非常清楚了
ID GeneID geneSymbol chr strand riExonStart_0base riExonEnd upstreamES upstreamEE downstreamES downstreamEE ID IJC_SAMPLE_1 SJC_SAMPLE_1 IJC_SAMPLE_2 SJC_SAMPLE_2 IncFormLen SkipFormLen PValue FDR IncLevel1 IncLevel2 IncLevelDifference