options(digits=2) covariances<-ability.cov$cov correlations<-cov2cor(covariances) #转化为相关矩阵 correlations

ability.cov提供了变量的协方差矩阵
cov2cor()函数将其转化为相关系数矩阵本
- 判断需提取的公共因子数
1 fa.parallel(correlations,n.obs=112,fa="both",n.iter=100,main="Scree plots with parallel analysis")
fa="both",则显示PCA和EFA两种线,如果选择fa="pc",则只会显示PCA的线,如果fa="fa",则只会显示因子分析的线。

若使用PCA方法,可能会选择一个成分或两个成分。当摇摆不定时,高估因子数通常比低估因子数的结果好,因为高估因子数一般较少曲解“真实”情况。
2.提取公共因子(因子旋转会更有效)
可使用fa()函数来提取因子,fa()函数的格式为:
fa(r,nfactors=,n.obs=,rotate=,scores=,fm)
r是相关系数矩阵或原始数据矩阵;
nfactors设定提取的因子数(默认为1);
n.obs是观测数(输入相关系数矩阵时需要填写);
rotate设定放置的方法(默认互变异数最小法);
scores设定是否计算因子得分(默认不计算);
fm设定因子化方法(默认极小残差法)。
与PCA不同,提取公共因子的方法很多,包括最大似然法(ml)、主轴迭代法(pa)、加权最小二乘法(wls)、广义加权最小二乘法(gls)和最小残差法(minres)。
- 未旋转的主轴迭代因子法
fa<-fa(correlations,nfactors=2,rotate="none",fm="pa")
- 正交旋转,因子分析的重点在于因子结构矩阵(变量与因子的相关系数)
fa.varimax<-fa(correlations,nfactors=2,rotate="varimax",fm="pa")
- 斜交旋转,因子分析会考虑三个矩阵:因子结构矩阵、因子模式矩阵和因子关联矩阵。虽然斜交方法更为复杂,但模型将更加符合真实数据。
fa.promax<-fa(correlations,nfactors=2,rotate="promax",fm="pa")

3.使用factor.plot()或fa.diagram()函数,可绘制正交或斜交结果的图形
factor.plot(fa.promax,labels=rownames(fa.promax$loadings))

fa.diagram(fa.promax,simple=TRUE)
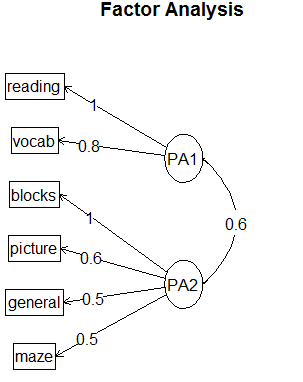
4.因子得分
EFA并不十分关注因子得分,在fa()函数中添加score=TRUE选项,便可轻松地得到因子得分。另外还可以得到得分系数(标准化的回归权重),它在返回对象的weights元素中。
fa.promax$weights
