Bioenv 分析通过 计算样本群落结构的距离矩阵和 环境因子的距离矩阵,计算两个距离之间的相关系数,挑选出最佳的环境因子组合;
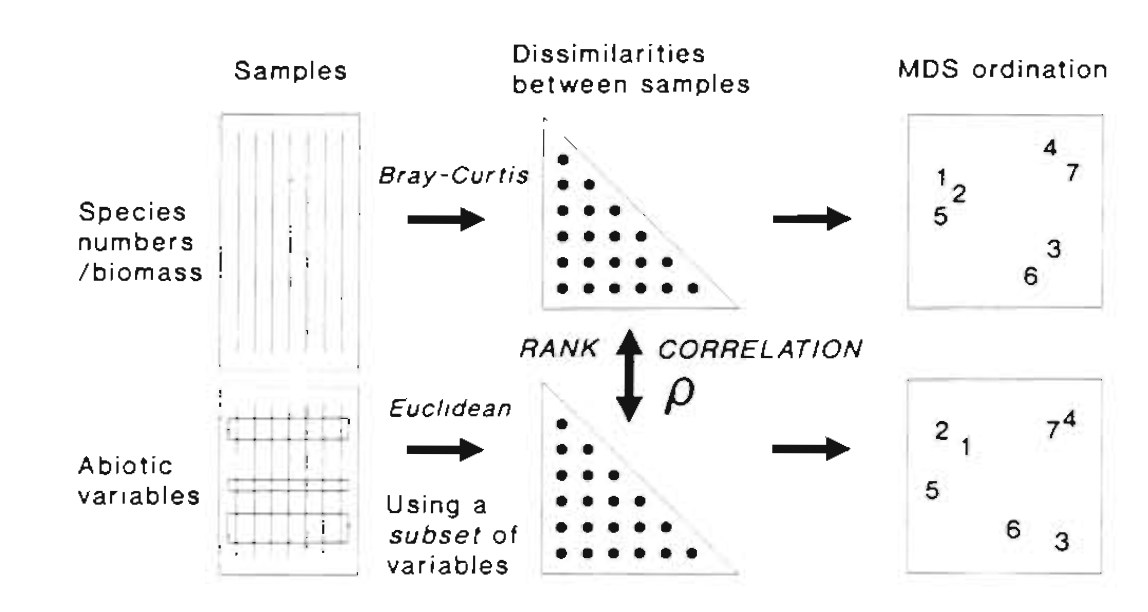
默认情况下,计算 群落结构的距离矩阵时, 使用 Bray-Curtis 距离; 计算环境因子的距离矩阵时,使用Euliodean 欧式距离, 计算相关性,则采用 spearman 相关系数;
示例用法:
> # The method is very slow for large number of possible subsets.
> # Therefore only 6 variables in this example.
> data(varespec)
> data(varechem)
> sol <- bioenv(wisconsin(varespec) ~ log(N) + P + K + Ca + pH + Al, varechem)
> sol
Call:
bioenv(formula = wisconsin(varespec) ~ log(N) + P + K + Ca + pH + Al, data = varechem)
Subset of environmental variables with best correlation to community data.
Correlations: spearman
Dissimilarities: bray
Metric: euclidean
Best model has 3 parameters (max. 6 allowed):
P Ca Al
with correlation 0.4004806
> summary(sol)
size correlation
P 1 0.2513
P Al 2 0.4004
P Ca Al 3 0.4005
P Ca pH Al 4 0.3619
log(N) P Ca pH Al 5 0.3216
log(N) P K Ca pH Al 6 0.2822
从上面的结果可以看出,最佳的环境因子的组合是P Ca Al , 因为其相关系数是最高的;
结果展现形式:
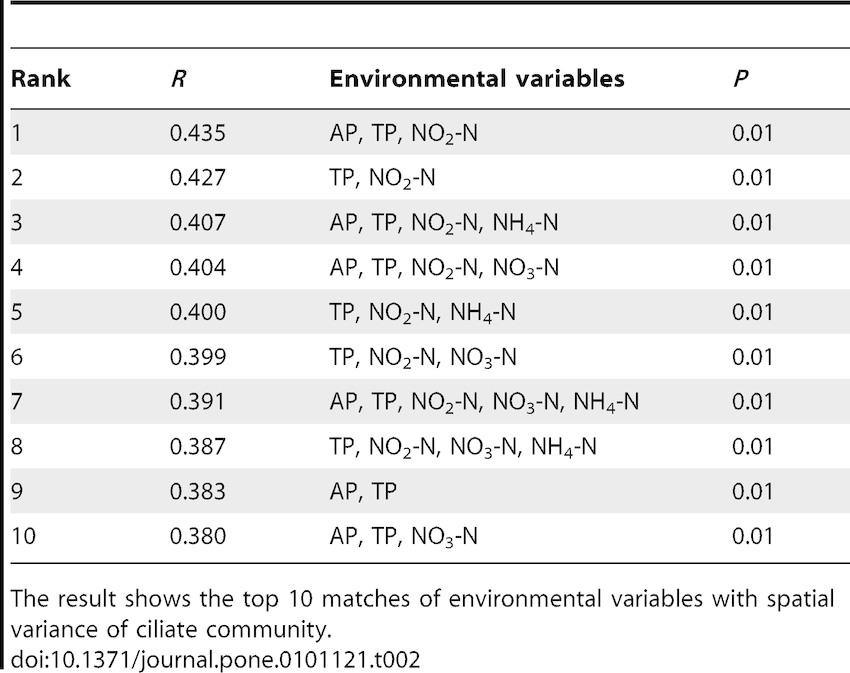
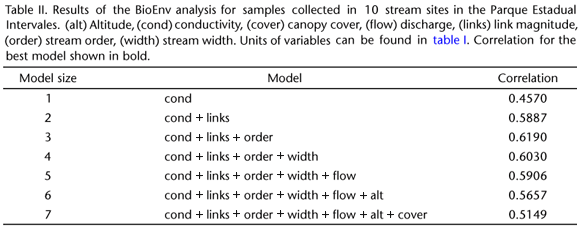
bioenv 分析的使用场景:
挑选合适的环境因子进行后续分析,当环境因子的个数较多时,可以使用bioenv 分析,挑选出相关性最高的环境因子的组合;
存在的问题:
当多个环境因子的组合相关系数差不多时,如果单纯的按照相关系数的值挑选1个最高的环境因子的组合,可能漏掉部分关键的环境因子;
参考资料:
https://www.rdocumentation.org/packages/vegan/versions/2.4-2/topics/bioenv
http://www.int-res.com/articles/meps/92/m092p205.pdf