LUNA16,全称Lung Nodule Analysis 16,是16年推出的一个肺部结节检测数据集,旨在作为评估各种CAD(computer aid detection计算机辅助检测系统)的banchmark,因为每个CAD都是基于自己的数据集,很难比较之间的性能优劣,这时候banchmark就很重要,在此之前比较知名的数据集就是Anode09了,不过这个数据集太小,训练集只有5个病例。
目前为止,比赛已经停止结果提交,不过数据集仍然可以下载,并且在leaderboard上有各路大神团队的比分及算法描述,可以自行查阅。
官方地址:https://luna16.grand-challenge.org
同时该数据集还有一个官方论文,Validation, comparison, and combination of algorithms for automatic detection of pulmonary nodules in computed tomography images: the LUNA16 challenge,本篇博客中主要内容也取于本论文。
论文地址:https://arxiv.org/abs/1612.08012
下面进入正题。主要从数据组成,比赛规则两方面介绍下这个数据集。
一 数据
LUNA16的数据来源于一个更大的数据集LIDC-IDRI,该数据集共有1018个CT扫描,也就是1018个病例,每个CT图像都有xml格式的标签文件,这个数据集的数据来源于7家不同的学术机构,所采用的扫描器及其相关参数都不尽相同,所以,1018个图像可以说分布不均,用论文中的话来说就是very heterogeneous。
LUNA16数据集将切片厚度(slice thickness)大于3mm的CT去除,同时将切片space不一致以及缺失部分切片的CT也去除,最后产生了888张CT,构成了LUNA16.这里要解释两件事,一,剔除3mm以上的CT是因为切片越薄效果越好,这不难理解,对肺部进行扫描,肯定是扫描得出的数据越多越好,想的极端点,如果整个肺部只有一张切片,这厚度绝对够了吧哈哈,那就连3D数据都没法获取了,更别说有效检测肺结节。二,切片space是啥,个人理解就是切片出来的数据是3D的(把一张张二维切片组合一起),有z,y,x三个维度,spacez指的就是切片厚度,spacey和spacex指的是每张切片的单个像素代表着实际宽高多少的肺部组织。所有的CT图像都以.mhd格式存储。
在LIDC-IDRI数据集中,有三种区域会标注出来,直径>3mm的结节,直径<3mm的结节以及非结节(但是肺部畸变区域),回到LUNA16,在888张CT中,共有36378个结节被标出(LIDC-IDRI标注的),在LUNA16中,只有直径>3mm的结节作为样本,直径<3mm的结节和非结节都不纳入进来,而直径<3mm的结节有11509个,非结节区域有19004个,这样还剩下36378-11509-19004=5765个,针对这5765个结节区域,若两个结节离的太近(此处太近的定义为中心距离小于半径之和,也就是相交了),则对两个结节进行合并,合并的中心和半径是该两个结节的均值,经过这样处理,还剩下2290个结节。由于标注的时候是四位专家一起标注,有些结节只有一位专家标注,有些有两位,最好的情况是四位都标注了,根据这个规则,分别有2290,1602,1186,777个结节由至少1,2,3,4位专家标注,LUNA16选取至少由三位专家标注的1186个结节作为最后要检测的区域,也就是我们做实验时下载的数据。
总结一下,数据筛选流程
(1)将直径>3mm的结节筛选出来,其它的不用,此时还有5765个结节
(2)将相近的结节融合,融合后还有2290个结节
(3)将三个以上专家标注的结节作为检测使用,共有1186个结节,也就是最后的实验数据。
(4)补充一点,不用的结节不是真的不用,2290-1186=1104个只有一位专家标注的结节,以及11509个直径<3mm的结节和19004个非结节合在一起作为irrelevant findings,这些区域,既不作为正样本也不作为负样本,所以如果你的算法检测出这些区域,不会处理为false positive,当然更不是true positive,直接无视之。
这里,不妨看一下实际上LUNA16的文件,共有10个子文件夹,subset0~subset9,这是为了做10折交叉验证,每个文件夹里都是病例,每个病例对应两个文件,文件名相同,后缀不同,其中.mhd文件存储着ct的基本信息,.raw文件存储着实际的ct数据,可以看到,ct文件还是挺大的,LUNA16足足一百多G,下载起来也挺耗时的。
二 比赛规则
比赛共有两项,一个是complete nodule detection,另一个是false positive reduction,前者要求实现一个完整的检测系统,后者要求对给定区域进行二分类,是否为结节。
我跑了一个github上的代码,只跑了任务一,所以任务二的比赛细节就不讲了。
具体而言,整个数据集是十份的,每份CT数相等,针对该数据集要执行10折交叉验证,总共可以分为四步
(1)取一份做测试集,其余九份做训练集
(2)在训练集上训练算法
(3)在测试集上测试,并生成结果文件
(4)完成10折交叉验证后,将所有结果融合为一份
最终提交文件要求为.csv格式,每行为一个标注,具体格式就是 image identifier,x,y,z,score,其中第一个代表某个CT,x,y,z为结节坐标,score为置信度。
对于提交的结节候选区,如何判断正负呢?如果提交的坐标位于结节半径范围内,则为正,也就是true positive,如果有多个候选区都与一个结节相关,则选取置信度最高的。若候选区检测出irrelevant nodules,则忽略掉,既不是正也不是负,剩下的候选区都可以归为false positive了。
有了正负样本的定义,就要进行评估,LUNA16采用的是FROC(Free-Response Receiver Operating Characteristic)准则,这一准则的定义及细节需要百度或者维基了解下。其实就是一张图,纵轴为召回率,横轴为FPs/scan,
召回率就是标记的结节你找出来几个,FPs/scan就是平均每个CT的FP数,注意是数,不是百分比。
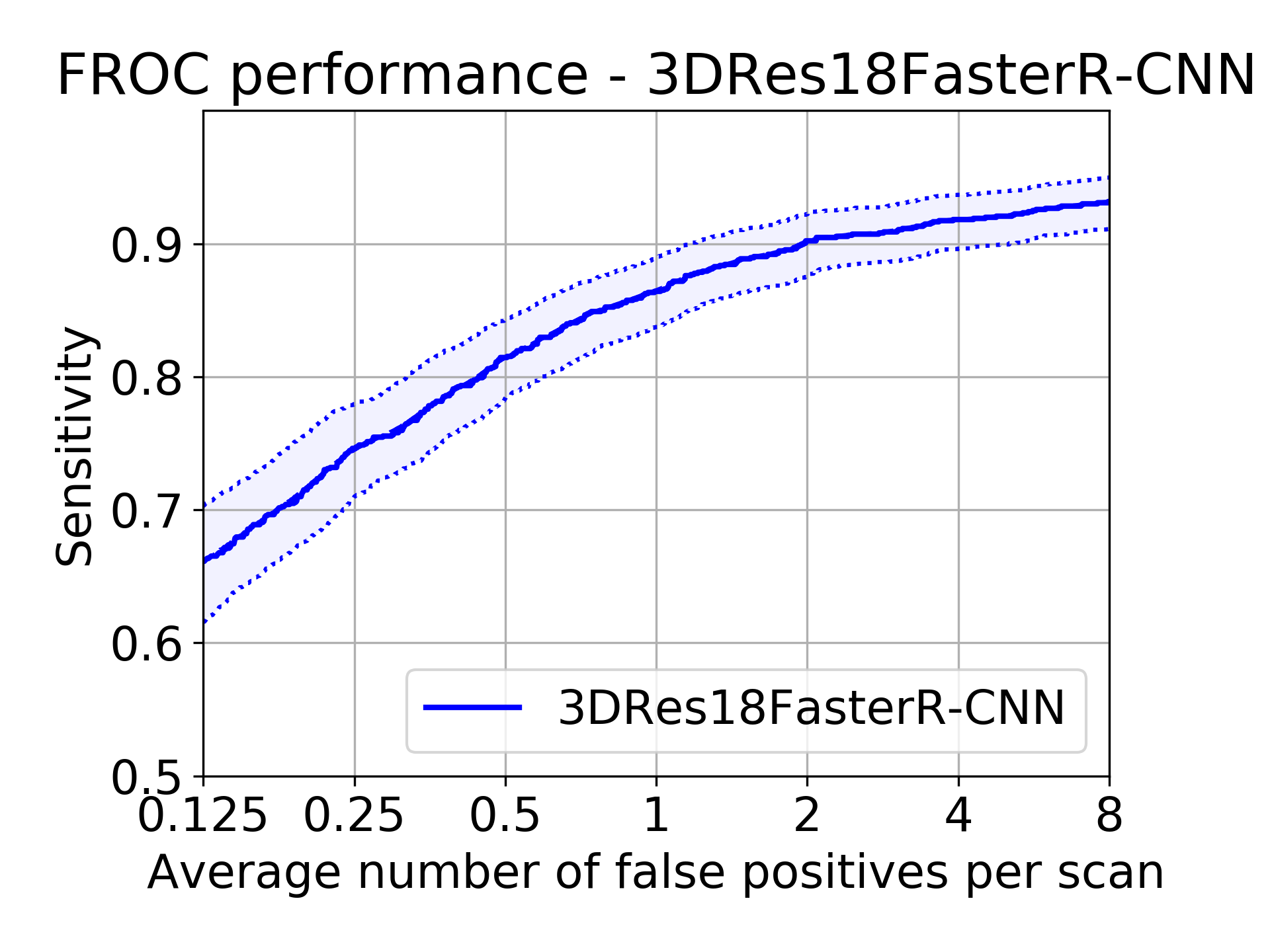
上图就是运行某个肺结节检测模型的FROC图,横轴明显是FP的数目。
最终的评比准则是0.125,0.25,0.5,1,2,4,8七个点召回率的平均值,论文中还提到bootstrap的方法,就是对测试集的每个CT进行重采样,最终就是有的CT可能出现多次,而有的CT可能没有,然后将采样得到的CT的候选区集合起来,计算FROC,不过bootstrap的置信度区间我没理解,有待考察。
目前任务一也就是检测的leaderboard已经被刷到了0.951的高分,来自平安科技,第二名0.950,来自健培公司,我跑的这个算法达到0.84,也不错,不过这前两名是真的恐怖,虚心学习之~