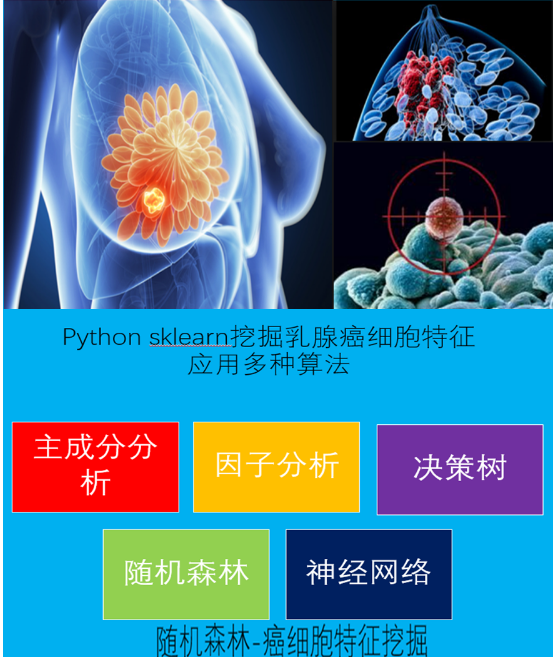
python机器学习-乳腺癌细胞挖掘(博主亲自录制视频)
https://study.163.com/course/introduction.htm?courseId=1005269003&utm_campaign=commission&utm_source=cp-400000000398149&utm_medium=share
支持向量机python代码和博客介绍
https://github.com/adashofdata/muffin-cupcake
YouTube介绍
https://www.youtube.com/watch?v=N1vOgolbjSc


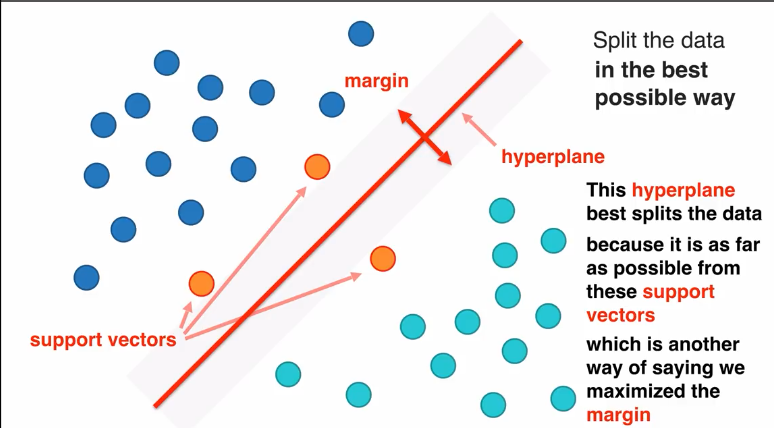
这是线性支持向量机,LSVM

margin
margin值越大越好,因为margin值越大,空间区分两组数据效果越好,margin值越小,空间区分两组数据效果越差
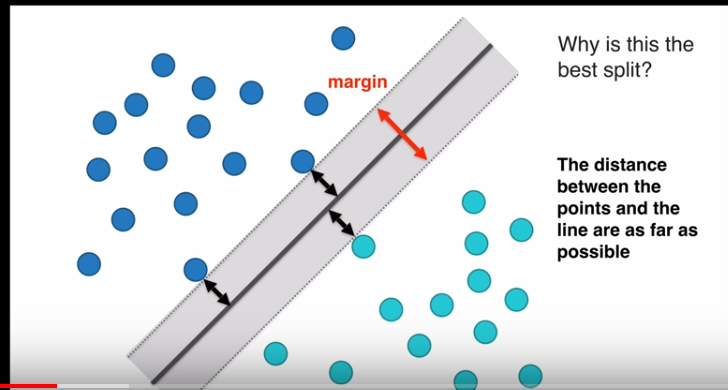
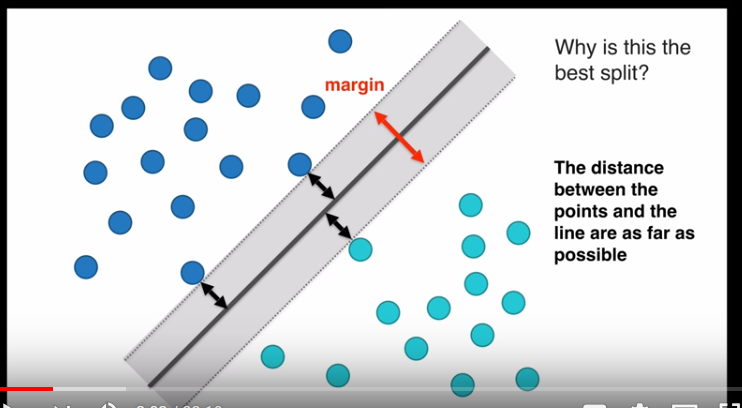
margin值最大的向量空间最好
lagrange multipliers拉格朗日乘数法是解决支持向量机margin最大值方法
在数学最优问题中,拉格朗日乘数法(以数学家约瑟夫·路易斯·拉格朗日命名)是一种寻找变量受一个或多个条件所限制的多元函数的极值的方法。这种方法将一个有n 个变量与k 个约束条件的最优化问题转换为一个有n + k个变量的方程组的极值问题,其变量不受任何约束。这种方法引入了一种新的标量未知数,即拉格朗日乘数:约束方程的梯度(gradient)的线性组合里每个向量的系数。 [1] 此方法的证明牵涉到偏微分,全微分或链法,从而找到能让设出的隐函数的微分为零的未知数的值。
支持向量优点
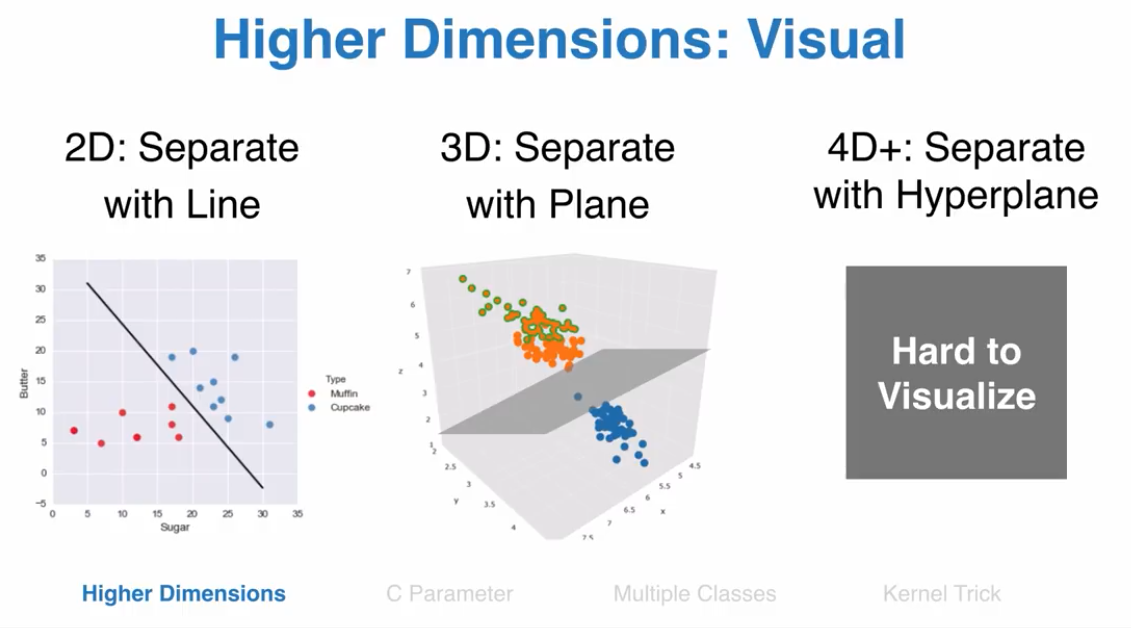
1.支持多维空间
2.不同核函数用于不同决策函数

支持多维空间

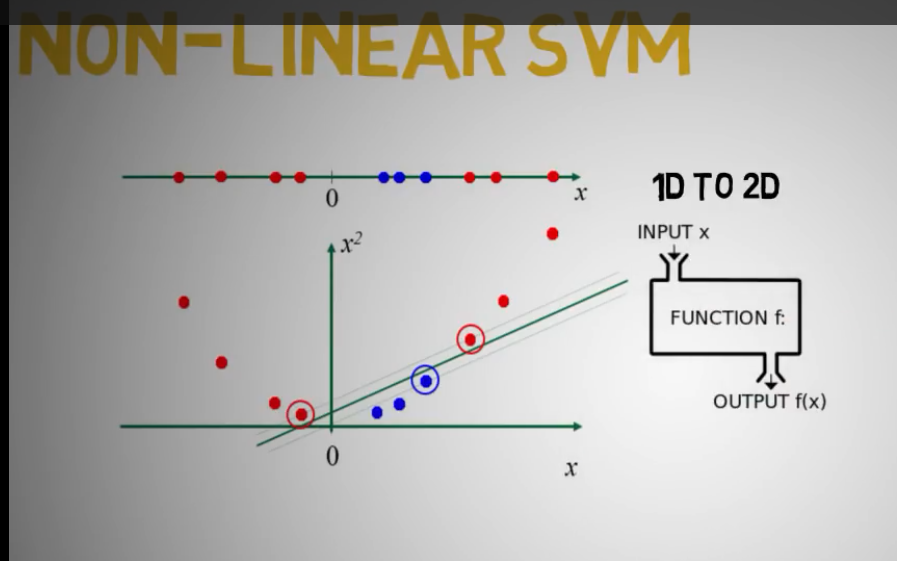
非线性SVM可以转换为多维空间支持向量机
支持向量缺点:
1.如果数据特征(维度)大于样本量,支持向量机表现很差
2.支持向量机不提供概率区间估计

优点:可处理多维度数据分类,小样本数据可以工作
缺点:找到准确的核函数和C参数,gamma参数需要很大计算量

优点:灵活,处理低维度和高维度数据,高维度数据小样本量表现良好
缺点:高维度,大样本表现较差,需要数据预处理和调参
很难监控和可视化
另外推荐算法:决策树和随机森林(方便可视化,监控,容易理解)

通过核函数,非线性空间可以转换为线性空间

支持向量应用积极广泛

python脚本应用
区分两种蛋糕,根据奶油和糖两种成分,首先数据可视化


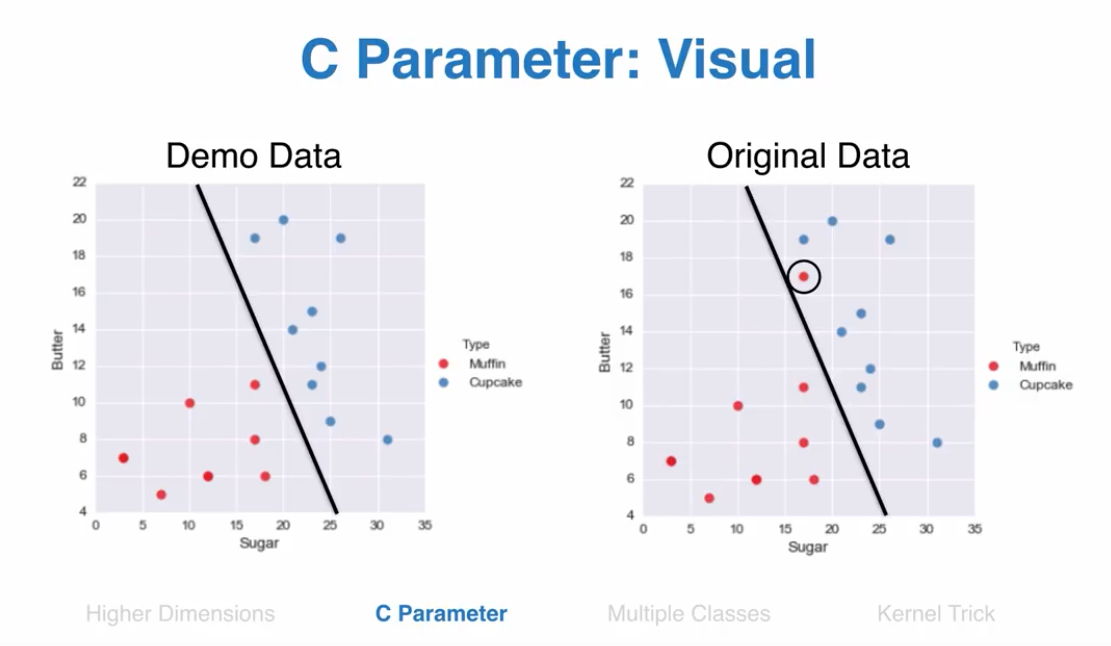
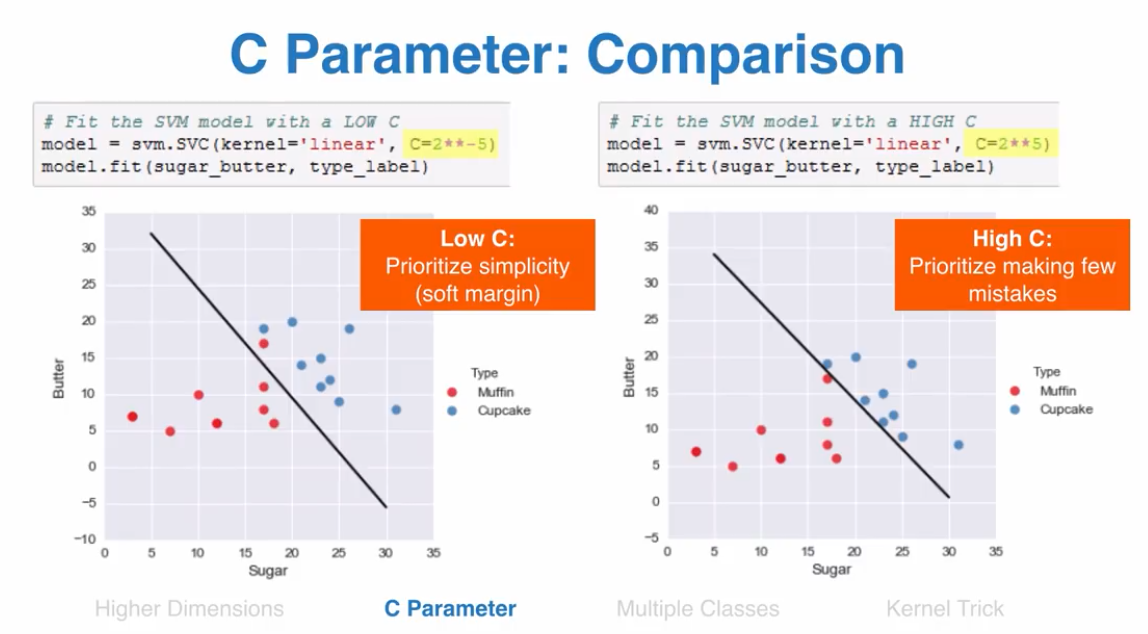
C 参数
C参数表示,你惩罚错误分类点的力度
sklearn默认C参数是1

C参数设置小,算法复杂度小,C参数高,追求错误率最低
支持多个分层

python代码实现分多类,decision_function_shape="ovr"


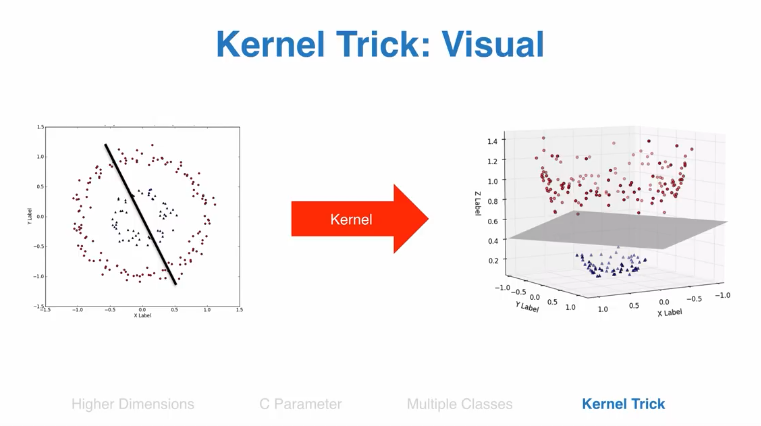
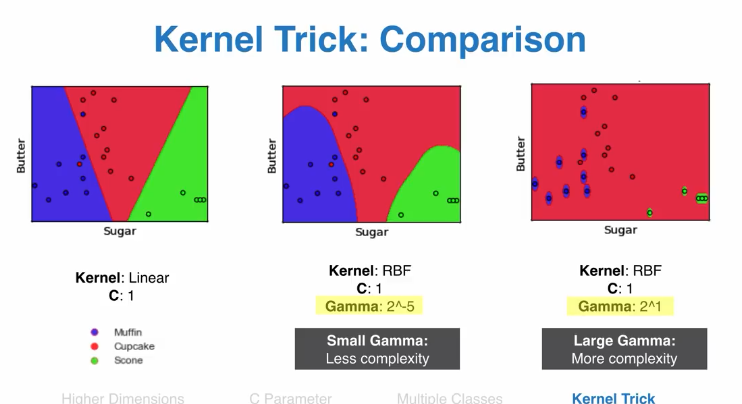
核函数
通过核函数,二维数据难以分类的可以转换为多维函数,然后分类

python代码kernel函数设置

gamma越高,复杂度越高
其它机器学习分类算法

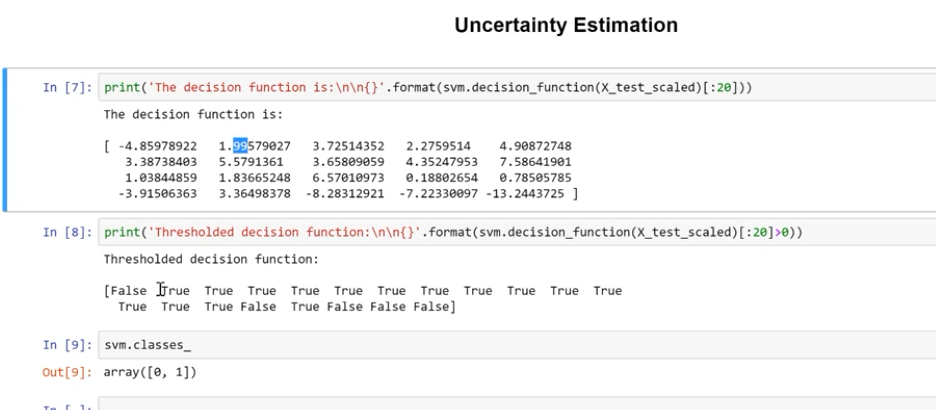
decision_function
SVM分割超平面的绘制与SVC.decision_function( )的功能
https://blog.csdn.net/qq_33039859/article/details/69810788?locationNum=3&fps=1
在李航老师的《统计学习方法》— 支持向量机那章有个例题:
样本点x1=(3,3),x2=(4,3),x3=(1,1),labels=(1,1,−1)x1=(3,3),x2=(4,3),x3=(1,1),labels=(1,1,−1),求分割超平面?
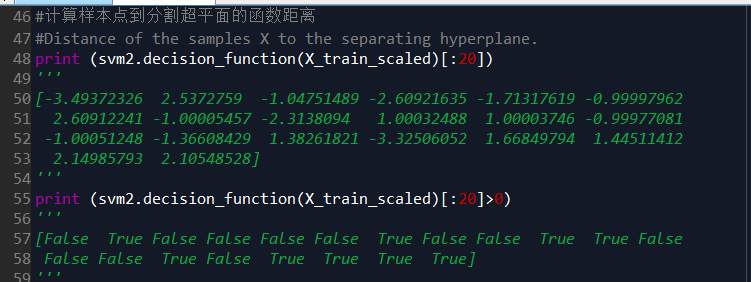
- 先说decision_function()的功能:计算样本点到分割超平面的函数距离。
- 没错,是函数距离(将几何距离,进行了归一化,具体看书)
- 将x1=(3,3),x2=(4,3),x3=(1,1),labels=(1,1,−1)x1=(3,3),x2=(4,3),x3=(1,1),labels=(1,1,−1)带入决策函数decision_function( ),也就是分割超平面12x1+12x2−212x1+12x2−2即可得到函数距离:1, 1.5, -1,其中1 和 -1 刚好在margin边缘上,x1,x3x1,x3也就是支持向量。
decision_function()的功能:计算样本点到分割超平面的函数距离,分割超平面一边数据是正数,一边是负数,如果是二分类,正数代表一类,负数代表另一类
乳腺癌python脚本
此脚本包括参数设置,自动调优,数据规范化,概率计算,分类预测等等
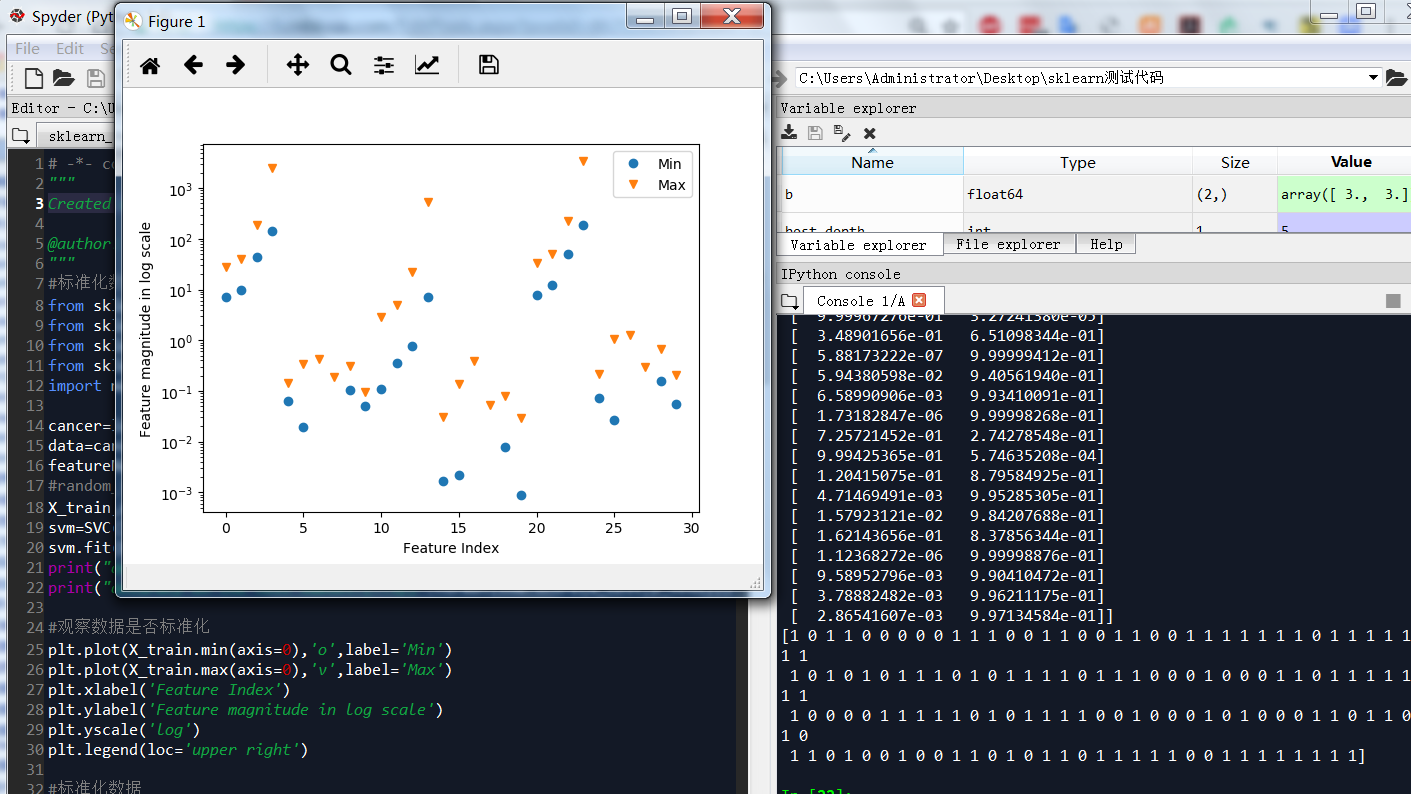
# -*- coding: utf-8 -*- """ Created on Fri Mar 30 21:57:29 2018 @author: Toby
项目合作联系QQ:231469242 """ #标准化数据 from sklearn import preprocessing from sklearn.svm import SVC from sklearn.datasets import load_breast_cancer from sklearn.model_selection import train_test_split import matplotlib.pyplot as plt cancer=load_breast_cancer() data=cancer.data featureNames=cancer.feature_names #random_state 相当于随机数种子 X_train,x_test,y_train,y_test=train_test_split(cancer.data,cancer.target,stratify=cancer.target,random_state=42) svm=SVC() svm.fit(X_train,y_train) print("accuracy on the training subset:{:.3f}".format(svm.score(X_train,y_train))) print("accuracy on the test subset:{:.3f}".format(svm.score(x_test,y_test))) #观察数据是否标准化 plt.plot(X_train.min(axis=0),'o',label='Min') plt.plot(X_train.max(axis=0),'v',label='Max') plt.xlabel('Feature Index') plt.ylabel('Feature magnitude in log scale') plt.yscale('log') plt.legend(loc='upper right') #标准化数据 X_train_scaled = preprocessing.scale(X_train) x_test_scaled = preprocessing.scale(x_test) svm1=SVC() svm1.fit(X_train_scaled,y_train) print("accuracy on the scaled training subset:{:.3f}".format(svm1.score(X_train_scaled,y_train))) print("accuracy on the scaled test subset:{:.3f}".format(svm1.score(x_test_scaled,y_test))) #改变C参数,调优,kernel表示核函数,用于平面转换,probability表示是否需要计算概率 svm2=SVC(C=10,gamma="auto",kernel='rbf',probability=True) svm2.fit(X_train_scaled,y_train) print("after c parameter=10,accuracy on the scaled training subset:{:.3f}".format(svm2.score(X_train_scaled,y_train))) print("after c parameter=10,accuracy on the scaled test subset:{:.3f}".format(svm2.score(x_test_scaled,y_test))) #计算样本点到分割超平面的函数距离 print (svm2.decision_function(X_train_scaled)) print (svm2.decision_function(X_train_scaled)[:20]>0) #支持向量机分类 print(svm2.classes_) #malignant和bening概率计算,输出结果包括恶性概率和良性概率 print(svm2.predict_proba(x_test_scaled)) #判断数据属于哪一类,0或1表示 print(svm2.predict(x_test_scaled))
机器分类是如何抉择的?
机器决策有两种方式,第一种依靠decision_function,计算点到分割超平面距离。如果距离绝对值比较明显,说明超平面分割效果较好。
decision_function是计算点到分割超平面距离的函数,
点到平面距离大于0的属于一类,<0的属于一类
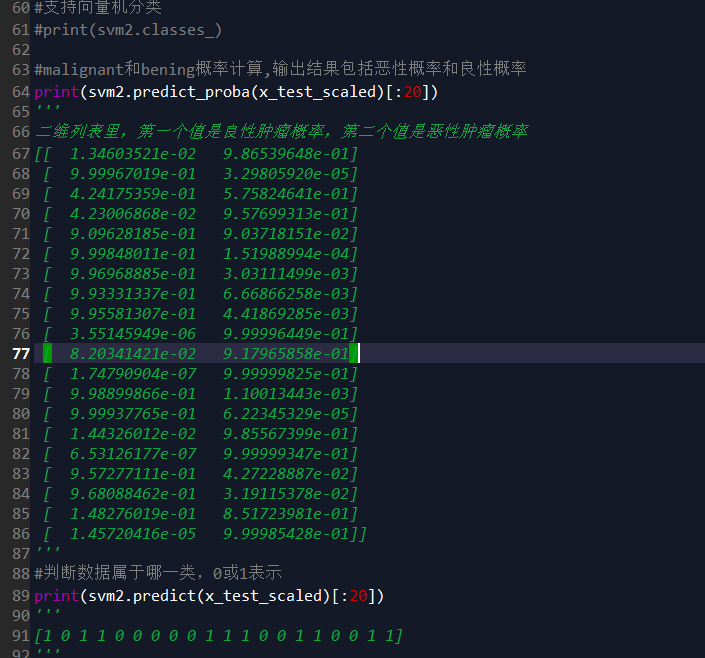
第二种方式是predict_proba是概率预测函数,概率大的值就属于对应分类
svm2.predict判断数据属于哪一类,0或1表示
SVM分类
SVM 支持向量机,在sklearn里面,有两种,SVC支持向量分类,用于分类问题,SVR,支持向量回归,用于回归问题。核方法
用于产生非线性分类边界。
linear,线性核,会产生线性分类边界,一般来说它的计算效率最高,而且需要数据最少。线性函数。
from sklearn import svm
svc = svm.SVC(kernel='linear')
svc.fit(X, y)poly,多项式核,会产生多项式分类边界。多项式函数。
svc = svm.SVC(kernel='poly',degree=4)
svc.fit(X, y)rbf,径向基函数,也就是高斯核,是根据与每一个支持向量的距离来决定分类边界的,它能映射到无限维,是最灵活的方法,但是也需要最多的数据。容易产生过拟合问题。指数函数。
svc = svm.SVC(kernel='rbf', gamma=1e2)多分类器
采用”one vs one”,在任意两个样本之间设计一个SVM,k个类别的样本设计k(k-1)/2个svm,当对一个未知样本进行分类时,最后得票最多的类别即为该未知样本的类别。
线性支持向量分类器(LinearSVC):相比于svm.SVC,使用了不同的算法,在某些数据集(比如稀疏数据集,文本挖掘)上运行得更快,对于多分类采用的就是”one vs all”的策略
svc=svm.LinearSVC(X,Y)支持向量
就是最靠近分离边界的样本点,它们是二分类问题中最具有代表性的点。支持向量的坐标可以通过方法support_vectors_来找到。
svc.support_vectors_[:, 0], svc.support_vectors_[:, 1]正则化
只考虑支持向量。使模型在处理样本特征的时候变得更加简单。
正则项可以通过调整系数C来决定
#大的C值:将会有较少的支持向量,决策边界是被大多数支持向量所决定。
svc = svm.SVC(kernel='linear', C=1e3)
#小的C值:将会有较多支持向量,决策边界=类别A的平均值-类别B的平均值
svc = svm.SVC(kernel='linear', C=1e-3)默认参数C=1,对于很多数据集,默认值就能工作的很好。
实践经验:对许多分类器来说,对样本正则化,采用标准差正则方法是非常重要的提升预测效果的手段。