我们可以假想有一个圆形的切割刀,随着半径的不同,我们可以得到许多分子的部分框架,然后按照这些框架的有无,对应的生成样子诸如0011011。。。的表达形式,这个东西就是该分子的分子指纹,如图所示。
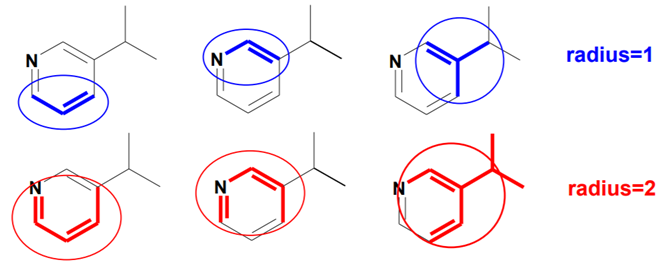
在Rdkit的2018.09版本的更新中,导入了新的工具rdkit.Chem.Draw。我们可以使用它来可视化morgan fingerprint等等除了Maccskey以外的分子指纹。
首先导入我们这次使用的library(请记得把rdkit更新到最新的版本):
import sys from IPython.display import SVG from rdkit import rdBase from rdkit import Chem from rdkit.Chem import AllChem, DataStructs from rdkit.Chem.Draw import DrawMorganBit, DrawMorganBits,DrawMorganEnv, IPythonConsole
导入目标分子&分子指纹化
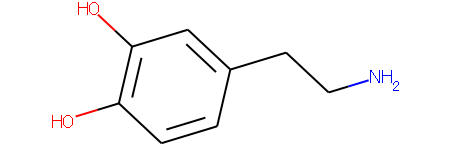
这次选择的目标分子是多巴胺。在pubchem里查到了它的Smiles之后,让我们将它的分子式可视化:
dopamine='C1=CC(=C(C=C1CCN)O)O' mol = Chem.MolFromSmiles(dopamine) mol
可以得到多巴胺的分子式:
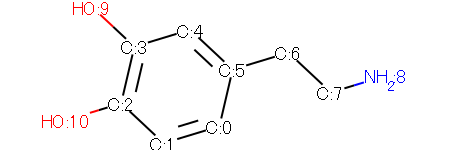
给多巴胺分子式添加原子序号
def add_atom_index(mol):
atoms = mol.GetNumAtoms()
for i in range( atoms ):
mol.GetAtomWithIdx(i).SetProp(
'molAtomMapNumber', str(mol.GetAtomWithIdx(i).GetIdx()))
return mol
# SetProp:给MOL添加新的物性信息
# mol.SetProp('prop_name',prop_val) str格式
# mol.SetIntProp('prop_name',prop_val) int格式
# mol.SetUnsignedProp('prop_name',prop_val) int格式 (正数)
# mol.SetDoubleProp('prop_name',prop_val) # float格式
# mol.SetBoolProp('prop_name',prop_val) # bool格式
add_atom_index(mol)
可以得到标记了原子序号的分子式:
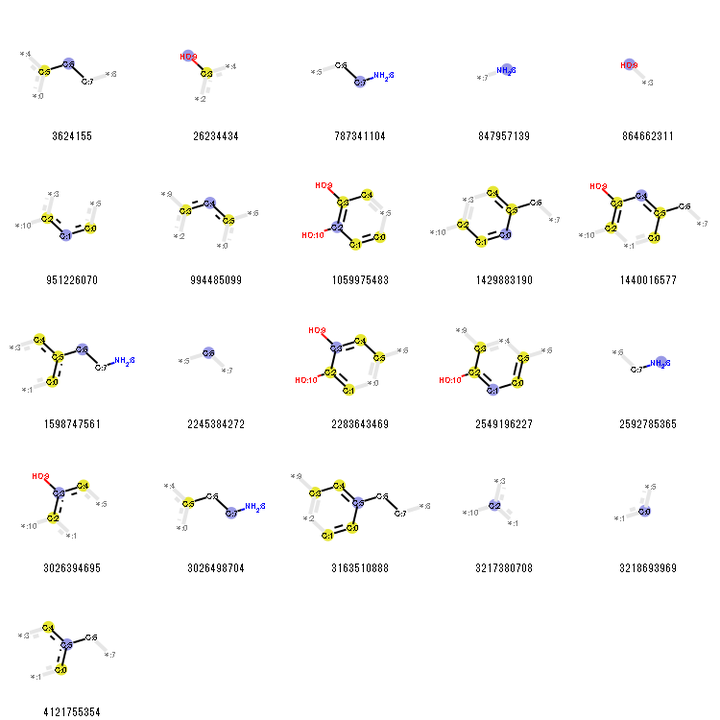
在最新的Rdkit的2018.09版本里,我们可以将Morgan fingerprint和Rdkit fingerprint两种分子指纹可视化。先将选项bitInfo设定为一个空的dict型的变数之后执行可视化。
ECFP_bitinfo = {}
ECFP = AllChem.GetMorganFingerprint(mol, radius=2, bitInfo=ECFP_bitinfo, useFeatures=False)
ECFP_tuples = [(mol, bit, ECFP_bitinfo) for bit in list(ECFP_bitinfo.keys())]
DrawMorganBits(ECFP2_tuples, molsPerRow=5, legends=list(map(str, list(ECFP2_bit_info.keys()))))
结果如下:
转自https://zhuanlan.zhihu.com/p/60796265
