K-medodis与K-means比较相似,但是K-medoids和K-means是有区别的,不一样的地方在于中心点的选取,在K-means中,我们将中心点取为当前cluster中所有数据点的平均值,在 K-medoids算法中,我们将从当前cluster 中选取这样一个点——它到其他所有(当前cluster中的)点的距离之和最小——作为中心点。K-medodis算法不容易受到那些由于误差之类的原因产生的脏数据的影响,但计算量显然要比K-means要大,一般只适合小数据量。 K-medoids 主要运用到了R语言中cluster包中的pam函数
K中心点聚类
- cluster::pam
- fpc::pamk
cluster::pam
Usage: pam(x, k, diss = inherits(x, "dist"), metric = "euclidean", medoids = NULL, stand = FALSE, cluster.only = FALSE, do.swap = TRUE, keep.diss = !diss && !cluster.only && n < 100, keep.data = !diss && !cluster.only, pamonce = FALSE, trace.lev = 0)
- x:聚类对象
- k: 是聚类个数 ( positive integer specifying the number of clusters, less than the number of observations)
示例代码
> newiris <- iris[,-5]
> library(cluster)
> kc <- pam(x=newiris,k=3)
> #kc$clustering
> #kc[1:length(kc)]
>
> table(iris$Species, kc$clustering)
1 2 3
setosa 50 0 0
versicolor 0 48 2
virginica 0 14 36
小结:
针对K-均值算法易受极值影响这一缺点的改进算法.在原理上的差异在于选择个类别中心点时不取样本均值点,而在类别内选取到其余样本距离之和最小的样本为中心。
fpc::pamk
相比于pam函数,可以给出参考的聚类个数, 参考 kmenas 与 kmeansrun
Usage: pamk(data,krange=2:10,criterion="asw", usepam=TRUE, scaling=FALSE, alpha=0.001, diss=inherits(data, "dist"), critout=FALSE, ns=10, seed=NULL, ...)
示例代码
newiris <- iris
newiris$Species <- NULL
library(fpc)
kc2 <- pamk(newiris,krang=1:5)
plot(pam(newiris, kc2$nc))
图例

fpc包还提供了另一个展示聚类分析的函数plotcluster(),值得一提的是,数据将被投影到不同的簇中
plotcluster(newiris,kc2$cluster)
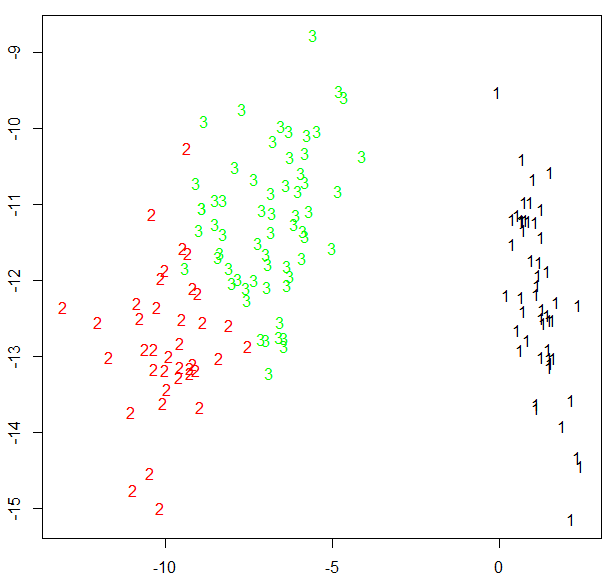
待验证:
为什么仅出现两个聚类?
参考资料: