原文链接:http://tecdat.cn/?p=14593
SIR模型定义
SIR模型是一种传播模型,是信息传播过程的抽象描述。
SIR模型是传染病模型中最经典的模型,其中S表示易感者,I表示感染者,R表示移除者。
S:Susceptible,易感者
I:Infective,感染者
R:Removal,移除者
SIR模型的应用
SIR模型应用于信息传播的研究。
传播过程大致如下:最初,所有的节点都处于易感染状态。然后,部分节点接触到信息后,变成感染状态,这些感染状态的节点试着去感染其他易感染状态的节点,或者进入恢复状态。感染一个节点即传递信息或者对某事的态度。恢复状态,即免疫,处于恢复状态的节点不再参与信息的传播。
SIR的微分方程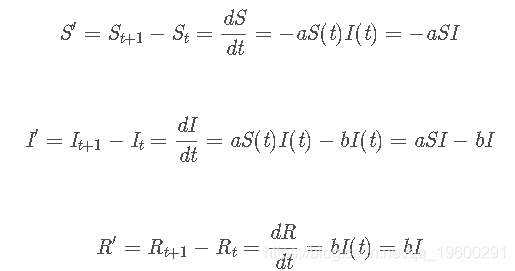
a为感染率、b恢复率
注意:
t为某个时刻,例如t=1,S(1)为第一天易感人群的人数。
无论t为什么时刻,总人数是不变的,即N(t)=S(t)+I(t)+R(t)。
人口总数总保持一个常数,即N(t)=K,不考虑人口的出生、死亡、迁移等因素。
这里介绍一个使用R模拟网络扩散的例子。
第一步,生成网络。
规则网
-
g =graph.tree(size, children =2); plot(g)
-
-
g =graph.star(size); plot(g)
-
-
g =graph.full(size); plot(g)
-
-
g =graph.ring(size); plot(g)
-
第二步,随机选取一个或n个随机种子。
-
# initiate the diffusers
-
-
seeds_num =1 diffusers =sample(V(g),seeds_num) ;
-
-
diffusers
-
-
## + 1/50 vertex:
-
-
## [1] 43
-
-
infected =list()
-
-
infected[[1]]=diffusers#
第三步,传染能力
在这个简单的例子中,每个节点的传染能力是0.5,即与其相连的节点以0.5的概率被其感染,每个节点的回复能力是0.5,即其以0.5的概率被其回复。在R中的实现是通过抛硬币的方式来实现的。
-
## [1] 0
-
显然,这很容易扩展到更一般的情况,比如节点的平均感染能力是0.128,那么可以这么写: 节点的平均回复能力是0.1,那么可以这么写
-
p =0.128
-
-
coins =c(rep(1, p*1000), rep(0,(1-p)*1000))
-
-
sample(coins, 1, replace=TRUE, prob=rep(1/n, n))
-
-
## [1] 0
-
-
n =length(coins2)
-
-
sample(coins2, 1, replace=TRUE, prob=rep(1/n, n))
-
-
## [1] 0
当然最重要的一步是要能按照“时间”更新网络节点被感染的信息。
-
keep =unlist(lapply(nearest_neighbors[,2], toss))
-
-
new_infected =as.numeric(as.character(nearest_neighbors[,1][keep >=1]))
-
-
diffusers =unique(c(as.numeric(diffusers), new_infected))
-
-
return(diffusers)}
-
-
set.seed(1);
开启扩散过程!
先看看S曲线吧:
为了可视化这个扩散的过程,我们用红色来标记被感染者。
-
# generate a palette#
-
-
plot(g, layout =layout.old)
-
-
set.seed(1)#
-
-
library(animation)# start the plot
-
-
m =1
如同在Netlogo里一样,我们可以把网络扩散与增长曲线同时展示出来:
-
set.seed(1)
-
-
# start the plot
-
-
m =1
-
-
p_cum=numeric(0)
-
-
h_cum=numeric(0)
-
-
i_cum=numeric(0)
-
-
while( m<50 ) {# start the plot
-
-
layout(matrix(c(1, 2, 1, 3), 2,2, byrow =TRUE), widths=c(3,1), heights=c(1, 1))
-
-
V(g)$color = "white"
-
-
V(g)$color[V(g)%in%infected[[m ]] ] = "red"
-
-
V(g)$color[V(g)%in%health[[m ]]] = "green"
-
-
if(m<=length(infected))
-
-
-
-
-
-
-
-
-
-
-
-
plot(pp~time, type ="h", ylab ="PDF", xlab ="Time",xlim =c(0,i), ylim =c(0,1), frame.plot =FALSE)
-
-
m =m +1
-
-
}

参考文献