
文章给出了一个可以对单样本识别在整个疾病进化过程中的关键基因的方法,这些关键基因很可能指导预后。
创新点:先前的动态模型寻找的方法往往不能对单样本进行分析,而是需要对不同时间结点构建的不同基因网络进行差异分析,寻找网络在不同样本中的突变点作为疾病的转折状态点,但是在真实的生物问题中往往测得的时间结点是有限的,甚至只有一个样本,为了解决这个问题,此文章利用了单样本的采样差异分析,定义样本内部基因-基因相似性网络随采样样本的不稳定性指数,并利用这一指数找寻关键基因。基于的假设是‘一组变量的强烈集体变动意味着状态发生改变’。
算法步骤:
1. 根据单样本数据构建基因相似性网络
2. 利用相似性网络定义样本内部基因-基因相似性网络随采样样本的不稳定性指数
3. 根据2的打分对所有基因进行降序排列,选出前k个作为关键基因
那么文章的关键就是怎么定义样本内部基因-基因相似性网络随采样样本的不稳定性指数。
首先先介绍一下多样本情况下如何定义网络发生了突变:

其中DNB(dynamic network biomarker)发生转折满足三个条件
1. 基因的标准差(方差开根号)变大,说明基因的表达情况出现了巨大波动
2. 基因和它的一节邻居之间的关系加强了,说明模块内部的链接加强了(集体变动)
3. 基因的一节邻居和基因对应的二阶邻居之间的关系减弱了,说明模块内部的链接减弱了(集体变动)
那么可以很自然的把网络变动指数定义为下面形式:
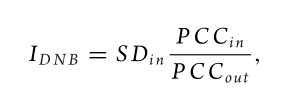
对于单样本的问题就是上面的所有都没办法通过跟reference样本对比得到,所以就要从单个样本上面定义,论文很巧妙的把问题转化为此基因在不同的细胞之间的表达情况是否稳定,即波动情况,这种波动情况可以由引入一个新的结点导致的相似性改变程度定义,公式如下:

PCC (pearson correlation coefficient)
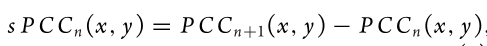
sPCC 微分形式表示波动情况

d这个细胞x基因的表达量减去x基因在该样本中的平均表达量,定义为sED(single-sample Expression Deviation)
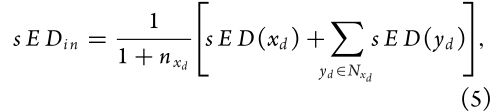
那么就有了基因x在模块(一节邻居)内部的波动程度,yd为x基因的所有一阶邻居,n_xd为一节邻居的个数
注意这里都是对于随机采样的一个细胞d来说的

模块内部链接的波动性就可以定义为上式(上式一定是递增的),模块与外部链接的波动性可以定义为下式:
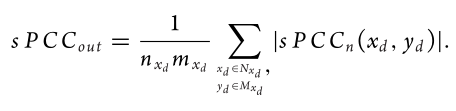
其中mdx为二阶邻居的个数,此式中,xd表示x的所有一阶邻居,yd表示所有二阶邻居。
那么就可以定义衡量基因内部稳定性的指标了:
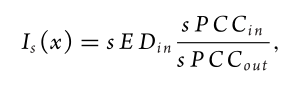
规则同多样本的三条,最后选出Is(x)前K个基因作为找到的关键基因。
构建SSN(single-sample network)可以使用sPCC作为边权重,也可以用PCC,或者其他的方法,都可以。用sPCC时,因为假设服从正态分布时候可以给出边权的置信度。(z-score)
对于多时间结点的动态网络分析见论文:
Detecting early-warning signals for
sudden deterioration of complex
diseases by dynamical network
biomarkers

主要是用到了非线性方程的特征值分解方法。