整个工程进展到这一步也算是不容易吧,但技术含量也不怎么高,中间乱起八糟的错误太烦人了,不管怎么样,现在面临了最大的困难吧,图像处理算法。算法确实不好弄啊,虽然以前整过,但都不是针对图像的。
现在的图像算法太多了,好像谁都在研究,没有一个统一的路线,看论文也是越看越糊涂,无奈之下还是自己好好学学吧,幸好队友以前也搞过,大家也都愿意参与进来了,很开心!
首先改变下策略吧,之前一直在linux中直接在QT中利用OpenCV库进行图像处理的尝试,但是效率太差了,每次想要结果,都要用板子,所以,现在改用OpenCV+vs2010现在PC上测试,直到满意了再复制到板子上进行测试。
换工具,得先配置啊,还好之前搞过,使用的人也多,所以比较顺利:参考博客
http://www.cnblogs.com/jamiechu/archive/2012/03/01/2376266.html
自己写程序,测试结果编译出错:LINK : fatal error LNK1123: 转换到 COFF 期间失败: 文件无效或损坏
百度了,http://www.cppleyuan.com/forum.php?mod=viewthread&tid=10226这个帖子不错,安装完补丁之后,编译生成解决方案成功。
测试了小程序,可以使用了。
针对自己的问题吧,现在的问题是月牙提取不出来,所以绞尽脑汁、千方百计要把这月牙给分离出来,找方法一个一个试吧。
话说加入OpenCV后编译又出现一个错误: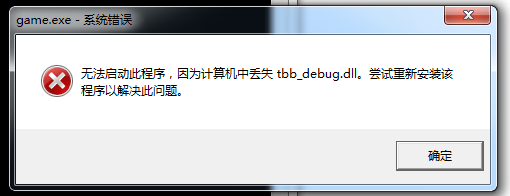
http://xuzhihong1987.blog.163.com/blog/static/2673158720122315223150/这个方法解决了。
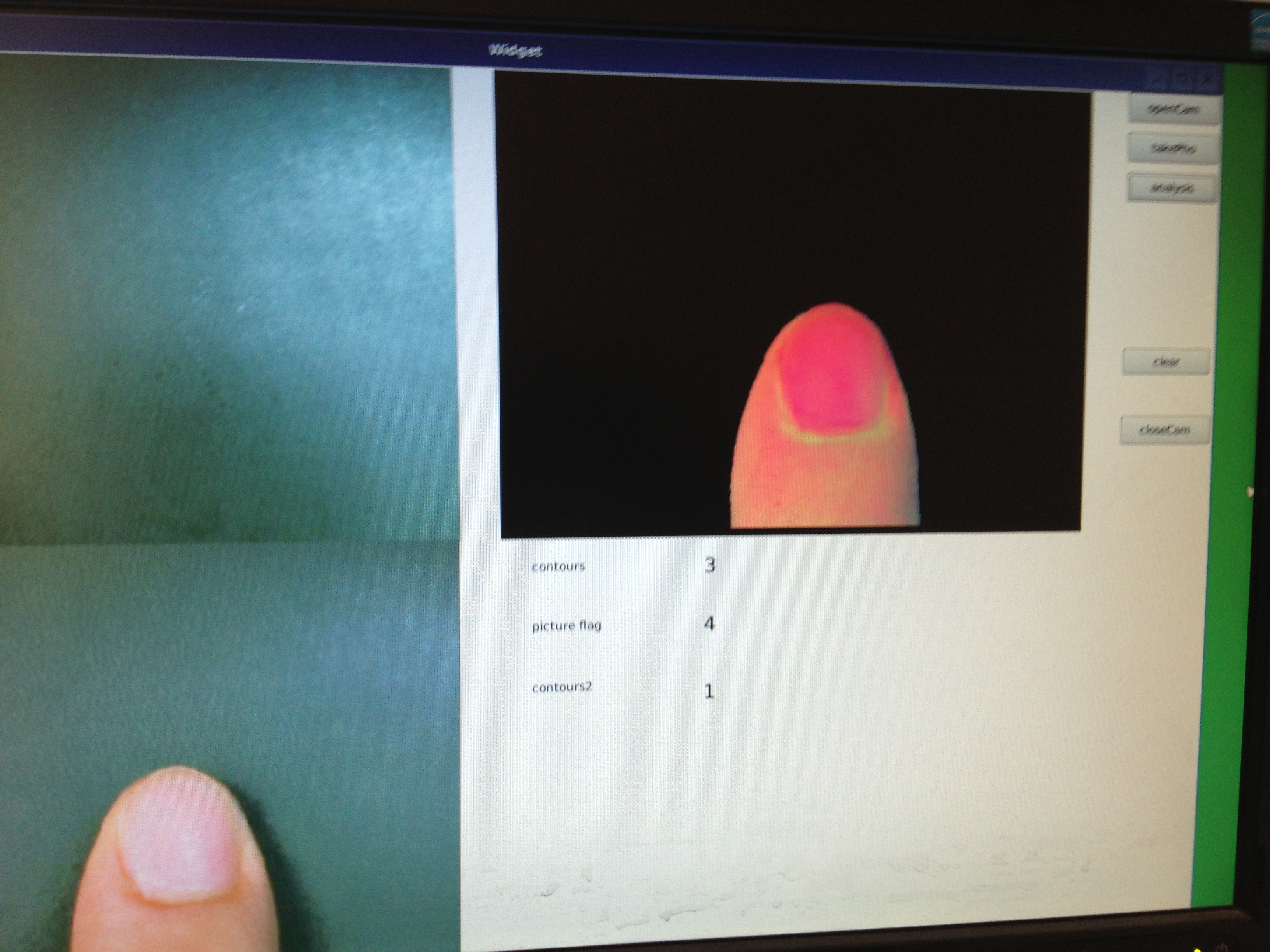
首先我们已经对图像进行了初步的分割,可以将指甲的轮廓提取出来,只是效果不是很理想。可以看到受光照影响明显,需要采取措施解决光照问题。如下图:
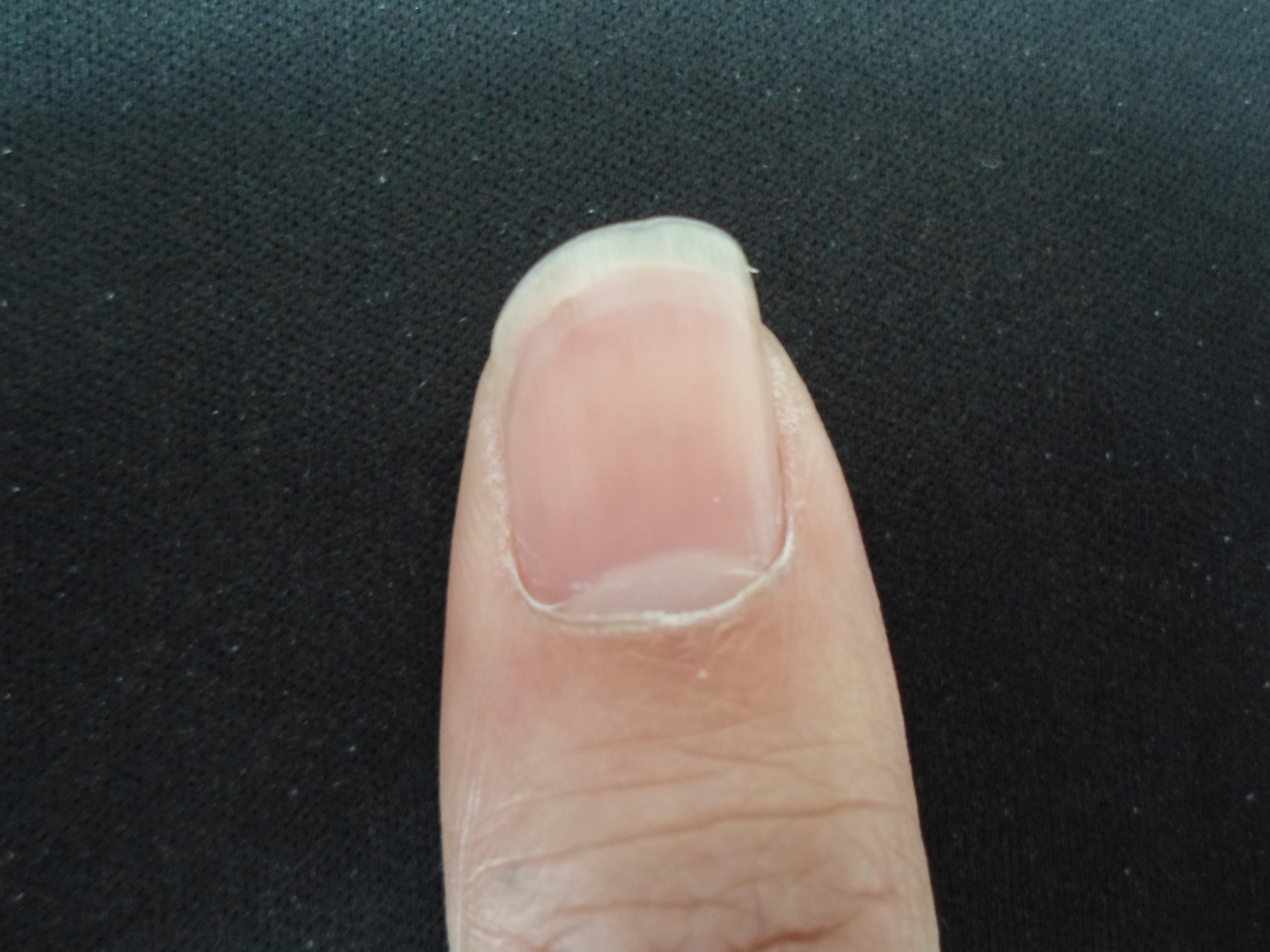
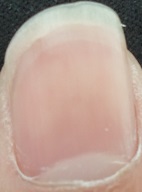


现在想要通过对扣取出来的图片进行进一步处理,也就是对第二幅图进行特征提取,从中找到我们所需的月牙,月牙和甲床面积比,月牙颜色,甲床颜色,甲床上是否有斑点、横纹、纵纹,从而为后面的医学诊断理论作依据。
既然基于灰度图像已经不能再有任何进展了,不如就用彩色图像分割吧。之前也探索过,发现还是有一定的效果的。
RGB颜色空间是图像处理中最基本、最常用、面向硬件的颜色空间。我们采集到的彩色图像,一般就是被分成R、G、B的成分加以保存的。然而,自然环境下获取的果实图像容易受自然光照、叶片遮挡和阴影等情况的影响,即对亮度比较敏感。而RGB颜色空间的分量与亮度密切相关,即只要亮度改变,3个分量都会随之相应地改变。所以,RGB颜色空间适合于显示系统,却并不适合于图像处理。
HSL 和 HSV(也叫HSB)是对RGB 色彩空间中点的两种有关系的表示,它们尝试描述比 RGB 更准确的感知颜色联系,并仍保持在计算上简单。
H指hue(色相)、S指saturation(饱和度)、L指lightness(亮度)、V指value(色调)、B指brightness(明度)。
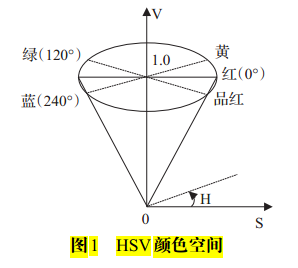 根据这幅图就能很好地理解HSV空间了。所以接下来就采用彩色图像进行分析看看效果。
根据这幅图就能很好地理解HSV空间了。所以接下来就采用彩色图像进行分析看看效果。
 ,貌似可以先将指甲抠出来。不管怎样,先试试。
,貌似可以先将指甲抠出来。不管怎样,先试试。
首先查论文,看到的好多是聚类方法,kmeans方法首先来,幸好OpenCV也有这函数,先来学学。
K-means算法是最为经典的基于划分的聚类方法,是十大经典数据挖掘算法之一。K-means算法的基本思想是:以空间中k个点为中心进行聚类,对最靠近他们的对象归类。通过迭代的方法,逐次更新各聚类中心的值,直至得到最好的聚类结果。
我的OpenCV的版本是2.3.1. 其中Kmean的实现在modulescoresrcmatrix.cpp里面,这里要推荐一个博客,讲得挺清楚:http://www.hongquan.me/?p=8
例子可以看:http://blog.csdn.net/xwu6614555/article/details/8568030
double cv::kmeans( InputArray _data, int K, InputOutputArray _bestLabels, TermCriteria criteria, int attempts, int flags, OutputArray _centers )
下面就是分析这个函数了。
_data: 这个就是你要处理的数据,例如是一个CvMat数据
K : 你需要最终生成的cluster的数量
_bestLabels: 当cv::kmeans执行完毕以后, _bestLabels里面储存的就是每一个对应的数据元素所在的cluster的index,这样你就可以更新你的数据,就是标记矩阵。
criteria: 这个东西是用来告诉cv::kmeans以一个什么样的停止条件来运行,例如criteria.epsilon = 0.01f;criteria.type = CV_TERMCRIT_EPS; 这个表示centers在两轮cluster运行以后的距离差,如果这个距离小于等于criteria.epsilon就停止返回当前得到的centers,否则继续
attempts: 最多尝试多少次,文档上说一般设置为2
flags: 这个主要是传递一些配置参数,例如 初始的时候使用user code给定的label –KMEANS_USE_INITIAL_LABELS,若使用kmeans++初始化算法– KMEANS_PP_CENTERS
_centers: 这个就是我们想要的结果了,该函数运行完毕以后,这个变量里面储存所有的center的数据,也就是你想要的东西了,引用例子的程序,稍作修改就拿来用了。
void kmeans_mat(const Mat& src_img,Mat& dst_img) { int width_src=src_img.cols; int height_src=src_img.rows; Mat samples=Mat::zeros(width_src*height_src,1,CV_32FC3);//创建样本矩阵,CV_32FC3代表32位浮点3通道(彩色图像) Mat clusters;//类别标记矩阵 int k=0; for (int i=0;i<height_src;i++) { for (int j=0;j<width_src;j++,k++) { //将像素点三通道的值按顺序排入样本矩阵 samples.at<Vec3f>(k,0)[0]=(float)src_img.at<Vec3b>(i,j)[0]; samples.at<Vec3f>(k,0)[1]=(float)src_img.at<Vec3b>(i,j)[1]; samples.at<Vec3f>(k,0)[2]=(float)src_img.at<Vec3b>(i,j)[2]; } } int nCuster=2;//聚类类别数,自己修改。 //聚类,KMEANS PP CENTERS Use kmeans++ center initialization by Arthur and Vassilvitskii kmeans(samples,nCuster,clusters,TermCriteria(CV_TERMCRIT_EPS+CV_TERMCRIT_ITER,10,1.0),2,KMEANS_PP_CENTERS); //显示聚类结果 if (dst_img.empty()) { dst_img=Mat::zeros(height_src,width_src,CV_8UC1); } k=0; int val=0; float step=255/(nCuster-1); for (int i=0;i<height_src;i++) { for (int j=0;j<width_src;j++,k++) { val=255-clusters.at<int>(k,0)*step;//int dst_img.at<uchar>(i,j)=val; } } }
来看看聚类结果先:
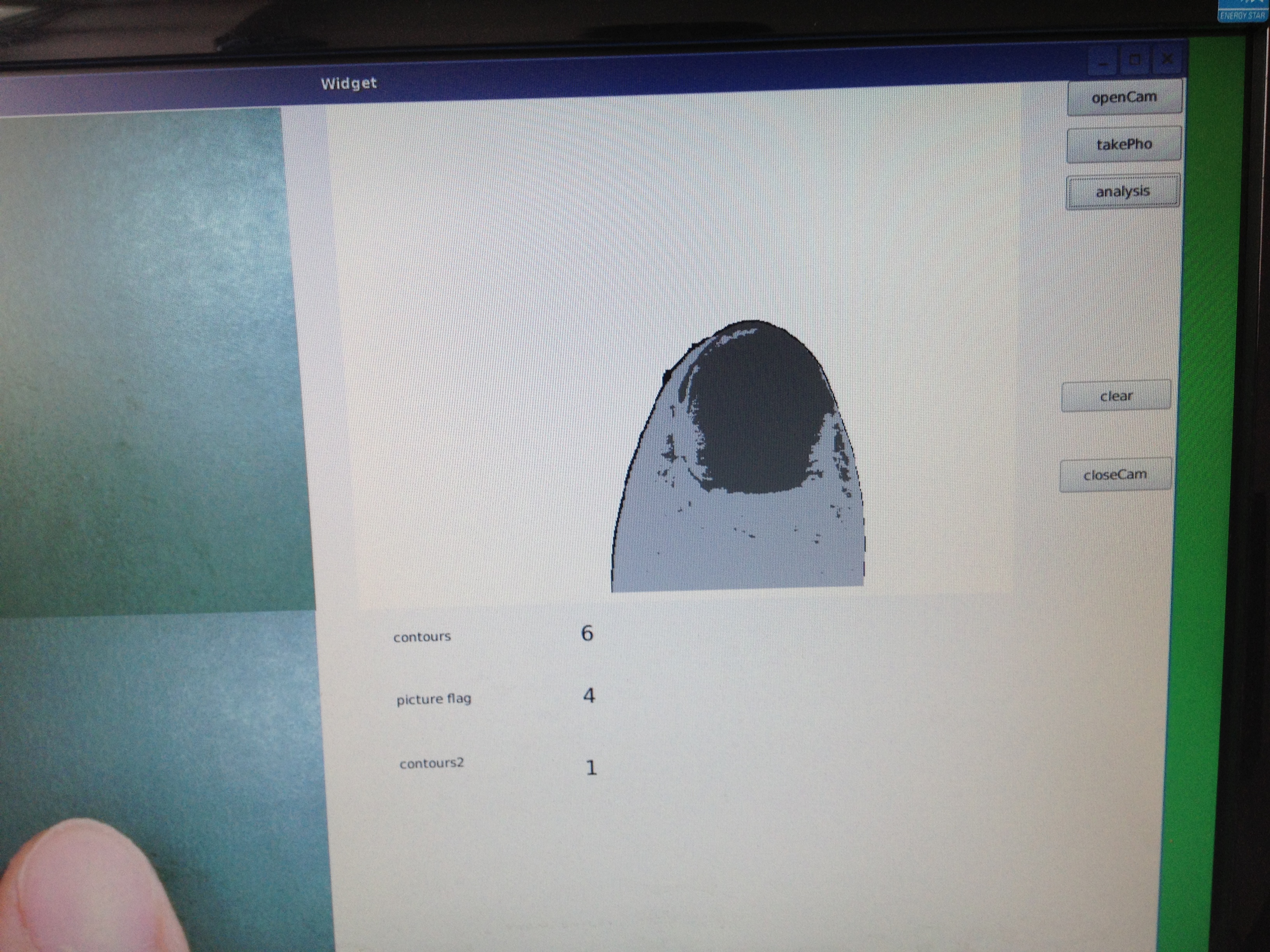 聚类效果还不错,只是比原本的指甲小了点,需要进一步修改,或者和前面的边缘提取相结合进行修正。而且聚类的数目需要人来控制。
聚类效果还不错,只是比原本的指甲小了点,需要进一步修改,或者和前面的边缘提取相结合进行修正。而且聚类的数目需要人来控制。
我在opencv的处理中总是遇到一个问题:Bad argument (Ukown array type) in cvarrToMat,后来发现是opencv库函数的使用问题,他有c的也有c++的,Mat一般是c++下的,所以用c的库函数会出现这个问题。
另外c版本中的保存图片为cvSaveImage()函数,c++版本中直接与matlab的相似,imwrite()函数。小插曲!