最近需要绘制基因调控网络,选择了python语言中比较popular的networkx包,先上目前结果图:
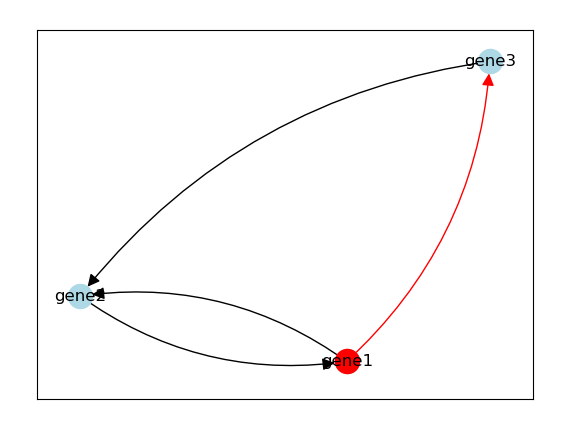
节点是基因或者其他实体,边代表调控关系,黑色的边代表抑制,红色的边代表激活,红色的节点代表自我激活,黑色的节点代表自我抑制。
绘制的关键点:边可以有不同的颜色,指向符号可以修改,可以绘制出自循环的边(self-loop)
本来抑制的边用截止符号-[更好,但是发现有时候看不清是来自哪条边的,所以就都改成箭头了,用颜色来区别关系;然后花了n久来找怎么绘制自循环的边,发现networkx的draw相关的方法 (基于matplotlib) 并不能显示(虽然我定义了节点n到节点n的边,但是结果不显示),查了一大堆发现确实是不支持的,好像说难实现还是咋回事...目前我已知的解决办法就是用另外一个绘图包graphviz,如该链接问题:https://stackoverflow.com/questions/49340520/matplotlib-and-networkx-drawing-a-self-loop-node
 (networkx的绘图包)
(networkx的绘图包)
我试了一下,确实可以,但效果没有之前好,所以决定换一种思路,用颜色代表自循环吧,用graphviz结果如下:
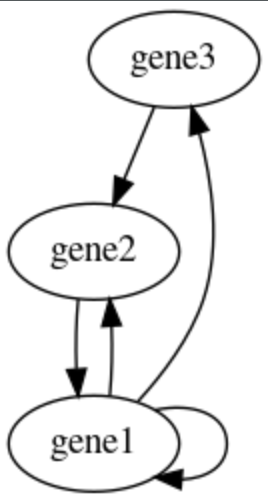
目前结果的代码如下:
1 import networkx as nx 2 import matplotlib.pyplot as plt 3 4 with open('grn_sample.txt', 'r', encoding='UTF-8') as fread: 5 cont = fread.readlines() 6 contLines = [] 7 genes = set() 8 G = nx.MultiDiGraph() 9 pEdges = [] 10 nEdges = [] 11 for line in cont: 12 tmp = line.strip().split(' ') 13 genes.add(tmp[0]) 14 genes.add(tmp[1]) 15 contLines.append(tmp) 16 genes = list(genes) 17 selfActGenes=set() 18 selfInhGenes=set() 19 G.add_nodes_from(genes) 20 for edge in contLines: 21 row = genes.index(edge[0]) 22 col = genes.index(edge[1]) 23 if edge[2] == '+': 24 pEdges.append((edge[0], edge[1])) 25 if row==col: 26 selfActGenes.add(edge[0]) 27 elif edge[2] == '-': 28 nEdges.append((edge[0], edge[1])) 29 if row==col: 30 selfInhGenes.add(edge[0]) 31 else: 32 print('Unsupported regulatory relationship.') 33 selfActGenes=list(selfActGenes) 34 selfInhGenes=list(selfInhGenes) 35 # show grn by network visualization 36 G.add_edges_from(pEdges) 37 G.add_edges_from(nEdges) 38 pos=nx.spring_layout(G) # 很重要! 39 nx.draw_networkx_nodes(G,pos,node_color='lightblue') # initial colors for all nodes 40 nx.draw_networkx_nodes(G, pos, nodelist=selfActGenes, node_color='red') # self-active nodes with red colors 41 nx.draw_networkx_nodes(G, pos, nodelist=selfInhGenes, node_color='black') # self-inhibited nodes with black colors 42 nx.draw_networkx_labels(G,pos) 43 nx.draw_networkx_edges(G,pos,edgelist=pEdges,edge_color='red',connectionstyle='arc3,rad=0.2',arrowsize=18) 44 nx.draw_networkx_edges(G, pos, edgelist=nEdges, edge_color='black',arrows=True,connectionstyle='arc3,rad=0.2',arrowsize=18) 45 plt.show()
读入文件如下(仅仅定义边):
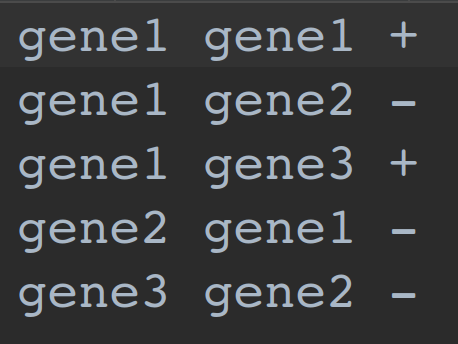
如果你需要使用截止符号,只需要在draw边的函数中定义参数arrowstyle,具体可以看官方文档,非常清晰: https://networkx.github.io/documentation/stable/reference/generated/networkx.drawing.nx_pylab.draw_networkx_edges.html
P.S. 对了,这次根据所需效果查询发现了直接看查询结果中图片结果的好处,可以通过图片直观的找到你要的效果所在的网页,节省很多时间。
