本次分享一篇今年2月才发表于Nature Methods的基于单细胞转录组数据推断基因调控网络的方法比较的文章,“Benchmarking algorithms for gene regulatory network inference from single-cell transcriptomic data[1]”,对应代码repo:https://github.com/murali-group/BEELINE。
摘要:他们对于从单细胞转录组数据中推断出的基因调控网络提出了一种系统级评价的最新算法。提出的评价框架叫做BEELINE。基于这些结果,他们可以对终端用户作出推荐。
随着单细胞测序技术的发展,一个中心的问题就是是否我们能够发现控制细胞分化和驱动细胞类类型转换的基因调控网络。在这样一个基因调控网络中,每条边连接一个转录因子到其调控的一个基因。理想情况下,这个边是从转录因子到目标基因的有向边,代表了直接而不是间接调控,并且对应于激活或者抑制状态。
他们提出了BEELINE,一个综合性的评价框架去评价基于单细胞基因表达数据的GRN推断技术的准确率,稳定性,有效性(图1,即整个框架的pipeline)。该框架包含了12种不同的GRN推断算法。以docker镜像的形式对每种方法提供了一个易于使用的标准接口。他还实现了许多种评价方法。
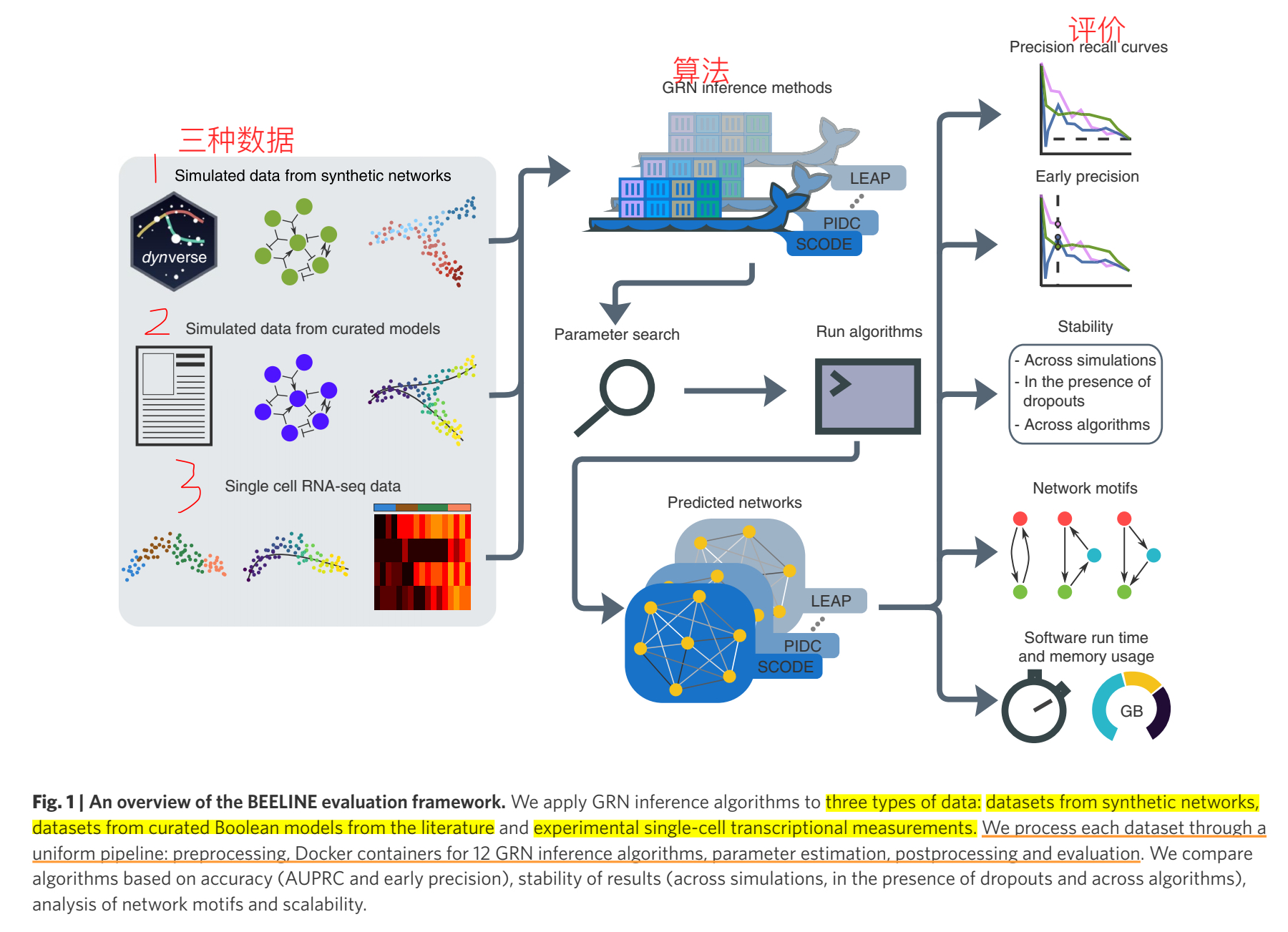
结果:
算法概览:他们使用这个框架在400个仿真数据集和5个实验数据集(人and鼠)去评价了这些方法。因为8个算法都需要伪时间顺序的细胞,所以他们的数据集都是关于细胞分化和发展的。也就是说细胞状态有一个有意义的时序过程。
然后他们结合三个数据集(来源于合成网络的数据集;来源于精选模型的数据集;实验数据集)进行的详细的论述,此处没有展开。
讨论:
图六总结了这些算法的性质和从这个研究中得到的一些看法观点。

尽管说对于不同类型的数据,算法的性能有较大差异,我们还是注意到了一些趋势。合成的网络比精选出来的模型更容易恢复。原因可能就是因为合成的网络有简单并且定义好的轨迹。对于精选出来的模型,每一个都有多条轨迹,我们发现不需要伪时间信息的方法表现得最好。针对布尔模型的方法表现的也很好,它推断出来的基因调控网络对实验数据集有比较好的准确率。然而,这些方法的总体性能都没有理想的好。并且经过分析,分析表明,这些算法可能都对准确的伪时间敏感。
基于这些观察,他们对用户在应用这些方法的时候做了具体的推荐:
(1)PIDC,GENIE3和GRNBoost2是可以选择的方法,因为他们在精选出来的模型和实验数据集上的准确率都比较好。
(2)PIDC,GENIE3在多次运行后有较好的稳定性。GRNBoost2对于dropout事件不敏感。因为这些方法不需要伪时间顺序的细胞,他们对伪时间计算中的错误是免疫的。随着伪时间推断质量提高,SINCERITIES可能变成一个一个好的选择,尤其是它也对dropout稳定。
(3)GENIE3和GRNBoost2有多线程的实现,他们和PIDC一样对小于等于2000个基因是有效的。
(4)他们的结果表明,加入更多的高可变基因或者是考虑所有关键的可变的转录因子,对结果是有提升的,尤其是EPR指标,对AUPRC指标没有影响。最近一篇最好的实践指导文章指出,使用1000到5000个高可变基因用于单细胞聚类或者分化表达任务,然而基因数量多可能会导致计算慢的问题。
虽然基因调控网络的推断已经研究了20年,但是我们的评价证明了在这个领域还存在一些具有挑战性的问题。一个可能的原因就是单细胞RNA测序技术可能没有提供足够的分辨率和表达的多样性。第二个可能就是一个固有的缺点来假设在表达模式和相应的调控交互之间的统计关系。最近的研究提出了多模态单细胞数据对于下一代基因调控网络推断算法可能是重要的。
BoolODE是我们分析的一个关键成分。可以考虑在研究新的基因调控网络推断方法的时候结合BEELINE和BoolODE一起使用。
方法:
首先简要的介绍了一下这12种算法的核心思想,这个在图6中已经有说明,我再用一个表格总结一下。
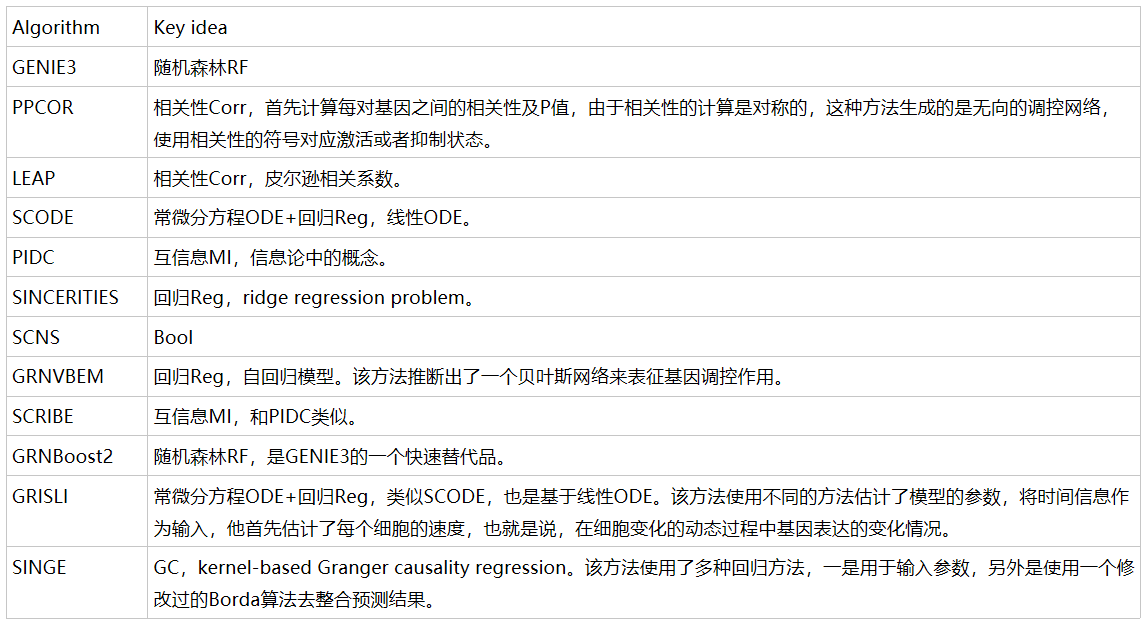
总结,大多数算法都需要有序的时间顺序的数据作为输入。除了PPCOR和PIDC,这里面几乎所有的算法的输出都是有向网络。只有五种方法的输出是有符号的。这些信息都反映在图6里面。
本篇文章中的方法都是不需要监督或者额外信息的,并且边都有权重。
BoolODE:
将布尔模型转换为ODE。GeneNetWeaver (GNW) 是一个被广泛使用的从GRN中仿真大量的转录数据 (bulk data) 的方法。当该工具被应用于单细胞分析的时候有限制。于是,他们提出了BoolODE,将Bool模型转换为随机微分方程SDEs。
对转录翻译建立了数学模型,然后仿真了这个SDE系统生成需要的基因表达数据。
BoolODE使用布尔模型创建了仿真数据集。这两个方法主要用于生成数据集。
后面是其他一些论文细节,最近会抽空补充完整!
Ref: Pratapa A, Jalihal A P, Law J N, et al. Benchmarking algorithms for gene regulatory network inference from single-cell transcriptomic data[J]. Nature Methods, 2020: 1-8.