PCA
主成分分析(Principal Component Analysis,PCA)是最常用的一种降维方法,通常用于高维数据集的探索与可视化,还可以用作数据压缩和预处理等。
目标
PCA可以把具有相关性的高维变量合成为线性无关的低维变量,称为主成分。主成分能够尽可能保留原始数据的信息。
相关术语
- 方差
- 协方差
- 协方差矩阵
- 特征向量和特征值



原理
矩阵的主成分就是其协方差矩阵对应的特征向量,按照对应的特征值大小进行排序,最大的特征值就是第一主成分,其次是第二主成分,以此类推。
推导过程详见周志华《机器学习》。

sklearn.decomposition.PCA
主要参数
- n_components:指定主成分的个数,即降维后数据的维度
- svd_solver :设置特征值分解的方法,默认为‘auto’,其他可选有‘full’, ‘arpack’, ‘randomized’。(可参考官网API,此处不详述)
应用:PCA实现高维数据可视化
目标
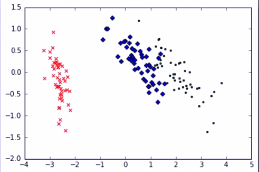
已知鸢尾花数据是4维的,共三类样本。使用PCA实现对鸢尾花数据进行降维,实现在二维平面上的可视化。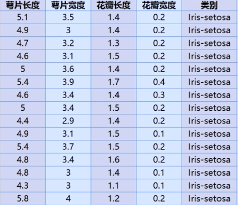

实例程序编写





1 import matplotlib.pyplot as plt 2 from sklearn.decomposition import PCA 3 from sklearn.datasets import load_iris 4 5 data = load_iris() 6 y = data.target 7 X = data.data 8 pca = PCA(n_components=2) 9 reduced_X = pca.fit_transform(X) 10 11 red_x, red_y = [], [] 12 blue_x, blue_y = [], [] 13 green_x, green_y = [], [] 14 15 for i in range(len(reduced_X)): 16 if y[i] == 0: 17 red_x.append(reduced_X[i][0]) 18 red_y.append(reduced_X[i][1]) 19 elif y[i] == 1: 20 blue_x.append(reduced_X[i][0]) 21 blue_y.append(reduced_X[i][1]) 22 else: 23 green_x.append(reduced_X[i][0]) 24 green_y.append(reduced_X[i][1]) 25 26 plt.scatter(red_x, red_y, c='r', marker='x') 27 plt.scatter(blue_x, blue_y, c='b', marker='D') 28 plt.scatter(green_x, green_y, c='g', marker='.') 29 plt.show()
NMF
非负矩阵分解(Non-negative Matrix Factorization ,NMF)是在矩阵中所有元素均为非负数约束条件之下的矩阵分解方法。NMF能够广泛应用于图像分析、文本挖掘和语音处理等领域。
基本思想:给定一个非负矩阵V,NMF能够找到一个非负矩阵W和一个非负矩阵H,使得矩阵W和H的乘积近似等于矩阵V中的值。
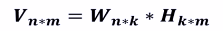

目标
第一种:最小化W矩阵H矩阵的乘积和原始矩阵之间的差别,目标函数如下:
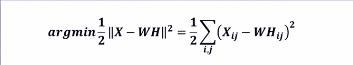 本公式基于欧式距离
本公式基于欧式距离
第二种:基于KL散度的优化目标,损失函数如下:

W和H的求法,严格来说,是它们的迭代算法。本课程不作详述,具体参考链接:

sklearn.decomposition.NMF
主要参数有:
- n_components:用于指定分解后矩阵的单个维度k;
- init:W矩阵和H矩阵的初始化方式,默认为‘nndsvdar’。其它参数参考官网API进行学习。
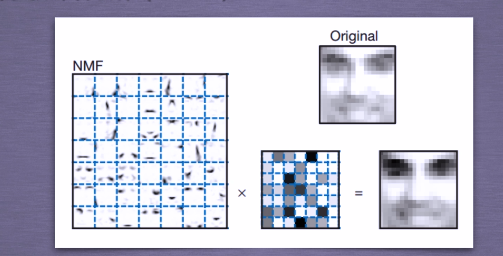
应用:NMF人脸数据特征提取
目标
已知Olivetti人脸数据共400个,每个数据是64*64大小。由于NMF分解得到的W矩阵相当于从原始矩阵中提取的特征,那么就可以使用NMF对400个人脸数据进行特征提取。


实例程序编写





1 from numpy.random import RandomState 2 import matplotlib.pyplot as plt 3 from sklearn.datasets import fetch_olivetti_faces 4 from sklearn import decomposition 5 6 7 n_row, n_col = 2, 3 8 n_components = n_row * n_col 9 image_shape = (64, 64) 10 11 12 ############################################################################### 13 # Load faces data 14 dataset = fetch_olivetti_faces(shuffle=True, random_state=RandomState(0)) 15 faces = dataset.data 16 17 ############################################################################### 18 def plot_gallery(title, images, n_col=n_col, n_row=n_row): 19 plt.figure(figsize=(2. * n_col, 2.26 * n_row)) 20 plt.suptitle(title, size=16) 21 22 for i, comp in enumerate(images): 23 plt.subplot(n_row, n_col, i + 1) 24 vmax = max(comp.max(), -comp.min()) 25 26 plt.imshow(comp.reshape(image_shape), cmap=plt.cm.gray, 27 interpolation='nearest', vmin=-vmax, vmax=vmax) 28 plt.xticks(()) 29 plt.yticks(()) 30 plt.subplots_adjust(0.01, 0.05, 0.99, 0.94, 0.04, 0.) 31 32 33 plot_gallery("First centered Olivetti faces", faces[:n_components]) 34 ############################################################################### 35 36 estimators = [ 37 ('Eigenfaces - PCA using randomized SVD', 38 decomposition.PCA(n_components=6,whiten=True)), 39 40 ('Non-negative components - NMF', 41 decomposition.NMF(n_components=6, init='nndsvda', tol=5e-3)) 42 ] 43 44 ############################################################################### 45 46 for name, estimator in estimators: 47 print("Extracting the top %d %s..." % (n_components, name)) 48 print(faces.shape) 49 estimator.fit(faces) 50 components_ = estimator.components_ 51 plot_gallery(name, components_[:n_components]) 52 53 plt.show()
