视频 https://www.bilibili.com/video/av7973580?from=search&seid=16993146754254492690
教材 Molecular biology of the gene 7th edition J.D. Watson et. al
DNA的复制,染色体组装
一、Replication Enzymes 复制酶
1、酶结构:
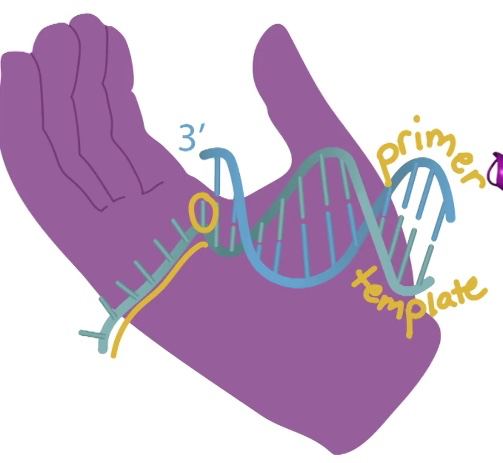
- 手状结构。thumb不动,而finger动。
- 手掌:①聚合作用。
- β折叠片层
- 2个二价金属离子(Mg2+orMn2+)
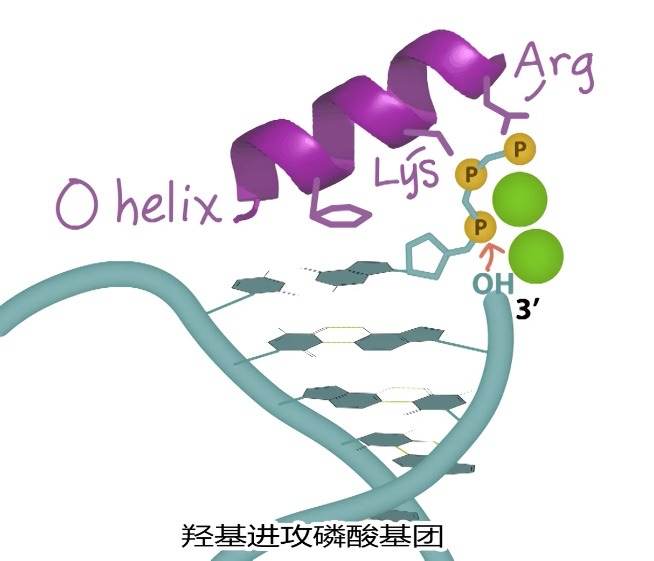
- 一个 降低3'-OH与H的亲和力,以利O-进行亲核攻击
- 一个 稳定焦磷酸
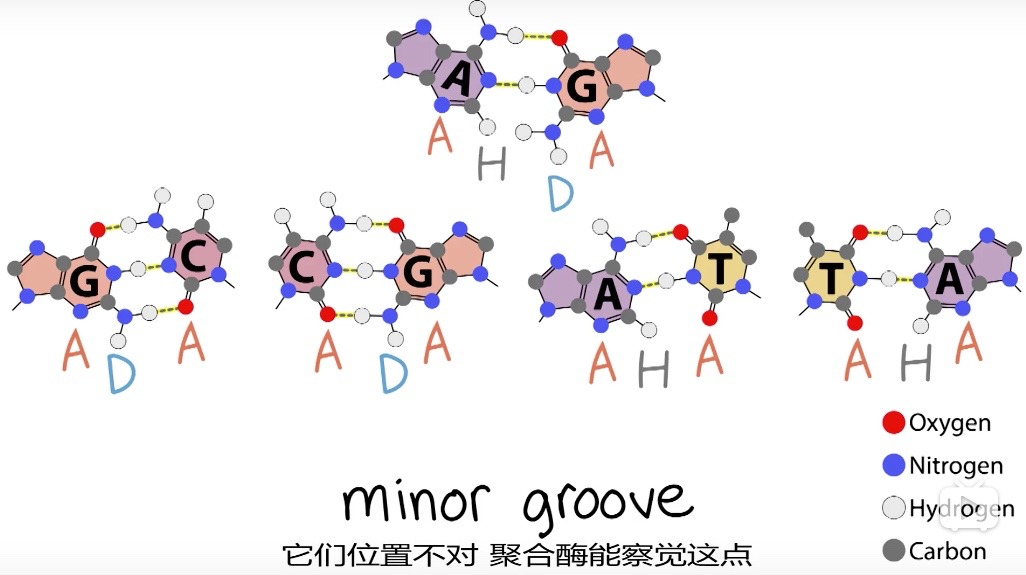
②错配检查:与小沟作用(无序列特异性)。错配时不能结合,并让部分链解开,以进入校对区
③外切酶活性位点
- 拇指,手指也与结构的固定有关 O-helix
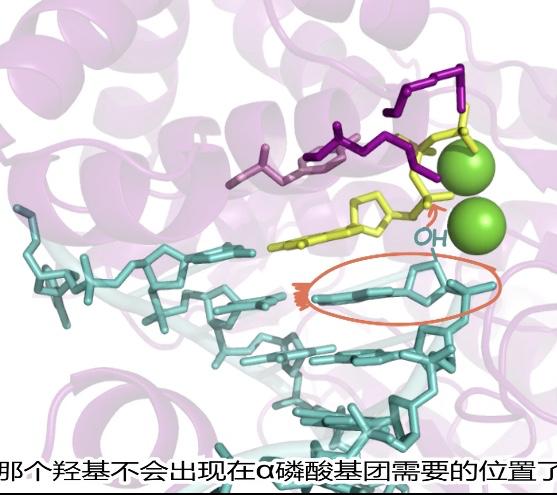
- 认的是底物位于正确的位点,错配和rNTP(2'-OH碰撞)都不能很好地位于正确位点
2、dNTP的掺入
1)dNTP被固定在特定位置(被O-helix压着防止水解)
- 碱基配对
- PPP与二价金属阳离子(Mg2+)作用:故金属螯合剂可以让聚合酶失活
- O helix和的酪氨酸和碱基的π-π相互作用
- Lys和Arg和磷酸基团的作用
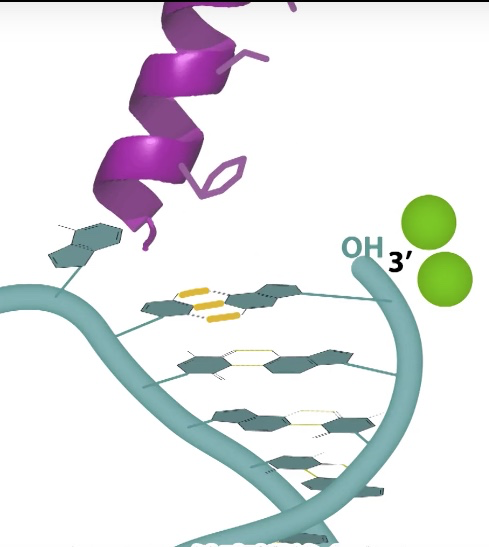
2)缩合,释放一个焦磷酸,O helix被释放
- 羟基攻击α磷酸
3)DNA移位,新的3‘ 位于活性中心
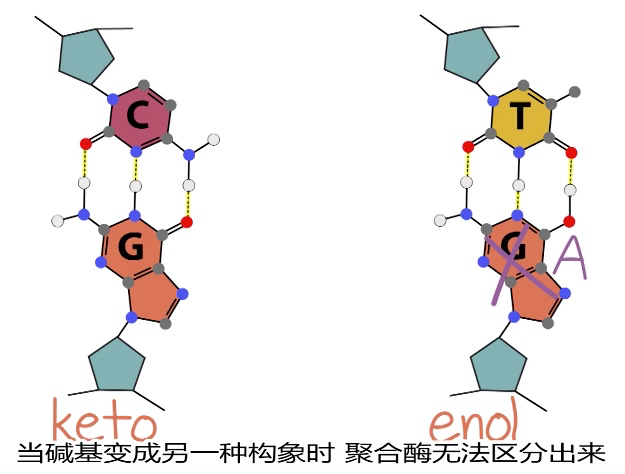
3、错配
每105bp会有一个错配
为何错配:
- 多为嘌呤-嘌呤,嘧啶-嘧啶间出错
- 碱基会异构:keto-enol 和 amino-imino
错配会减慢合成速度,在这时,聚合酶的外切活性位点会修复
- Proofreading exonuclease 外切酶 3’->5‘活性
- 同一酶的不同活性中心
- 错配时,将与外切中心亲和力高
- 这让错误率降低到 1mistake/107bp
二、Replication Fork 复制叉(原核为例)
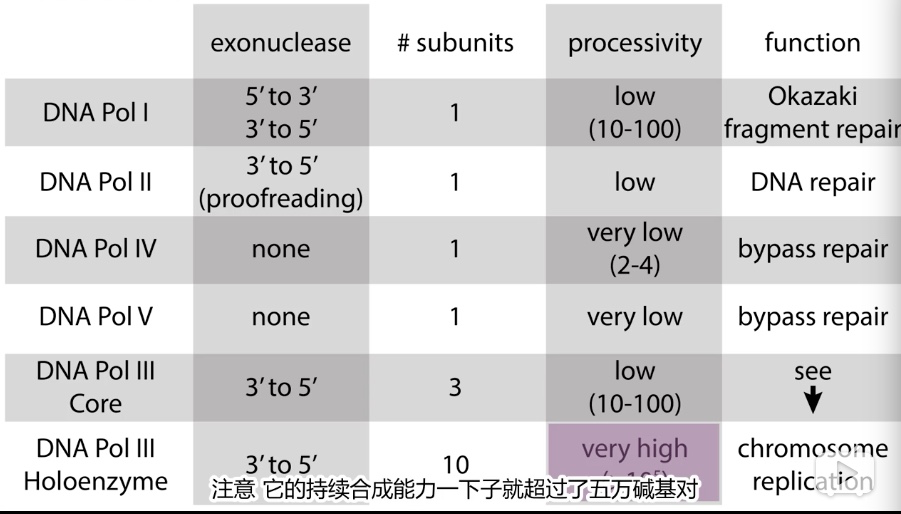
1、聚合酶
- 聚合酶I 5'->3' 3'->5'切, 短聚合
- 聚合酶III为核心酶的 全酶 3'->5'切 , 长聚合
- 其他 修复
2、DNA Primase 引物酶
- 合成RNA引物
- 要和helicase结合以提升活性,故只在复制叉处有活性
Question 引物酶持续合成能力测定?
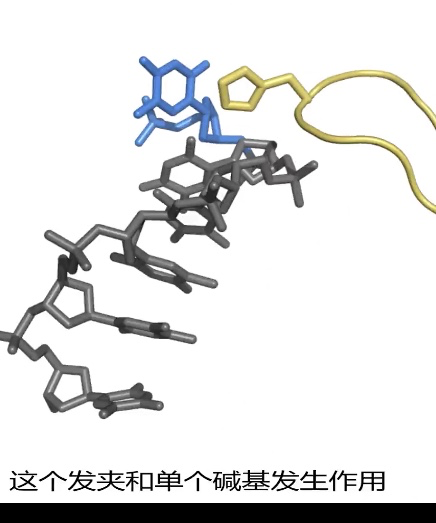
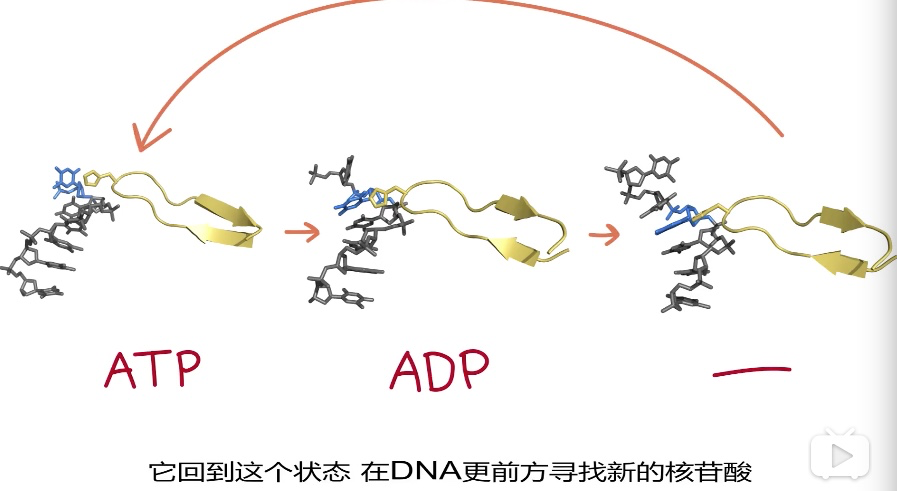
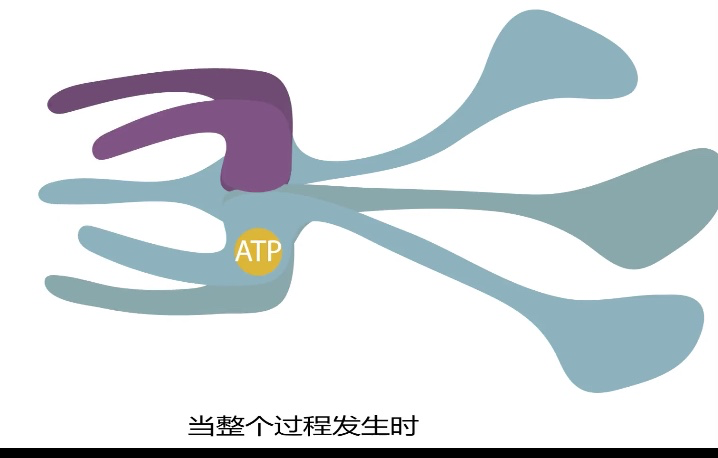
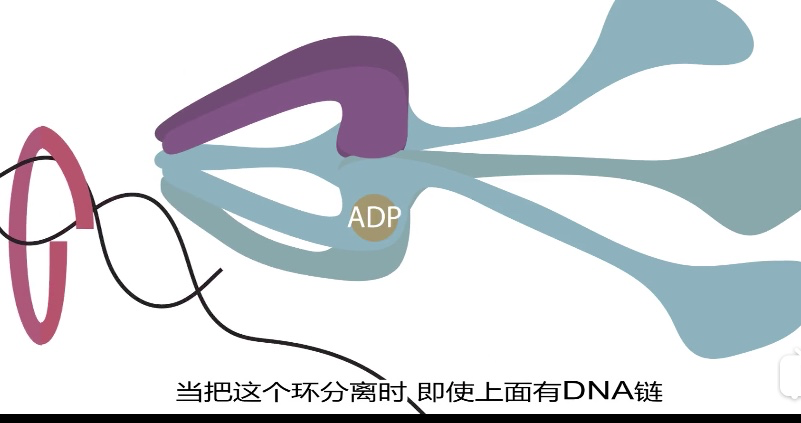
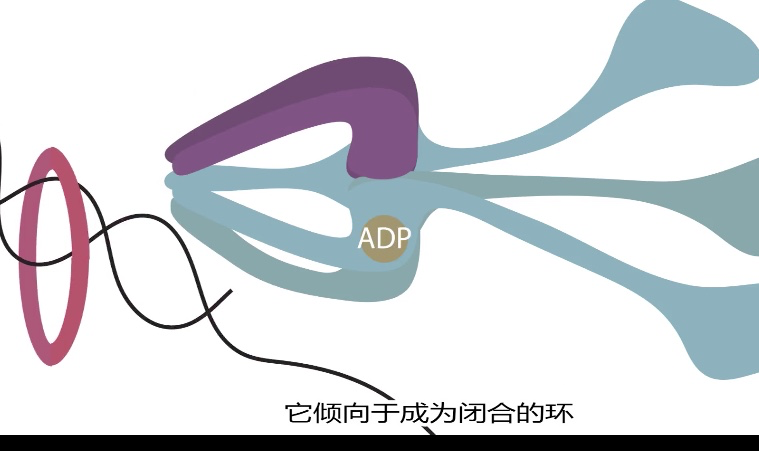
3、 DNA helicase 解旋酶
结合+ATP-->水解-ADP&Pi-->放开
| |
<-----向前移位--------
- 结合ATP后返回顶部,发夹结合碱基->随着ATP水解和释放->底部
- 原核中 环绕后随链
- 真核中环绕先导链
- Helicase是双向都能走的
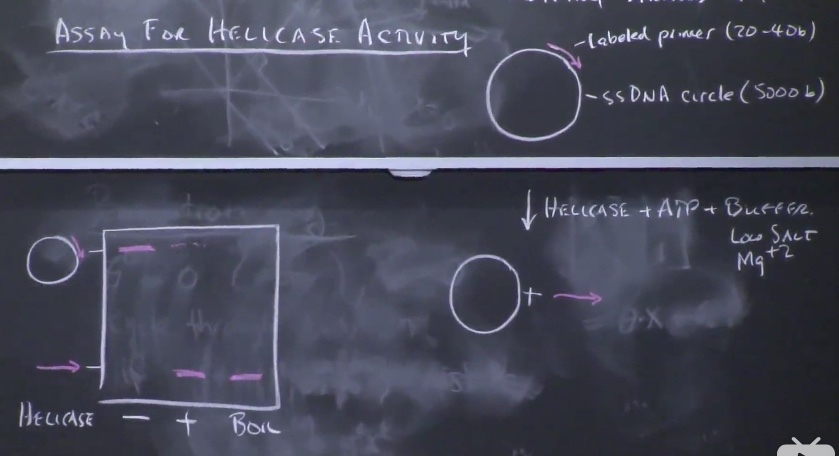
assay for helicase
- 本胶应非变性胶
- 如想看到所有DNA 溴乙锭染色
- 为何低盐助变性:高盐高电解质强度,掩盖了DNA双链负电。低盐,负电互斥
assay for helices polarity
用限制酶切段双链部分
4、SSB:协同结合的方式,结合ssDNA。与骨架静电作用,与碱基疏水堆积作用。
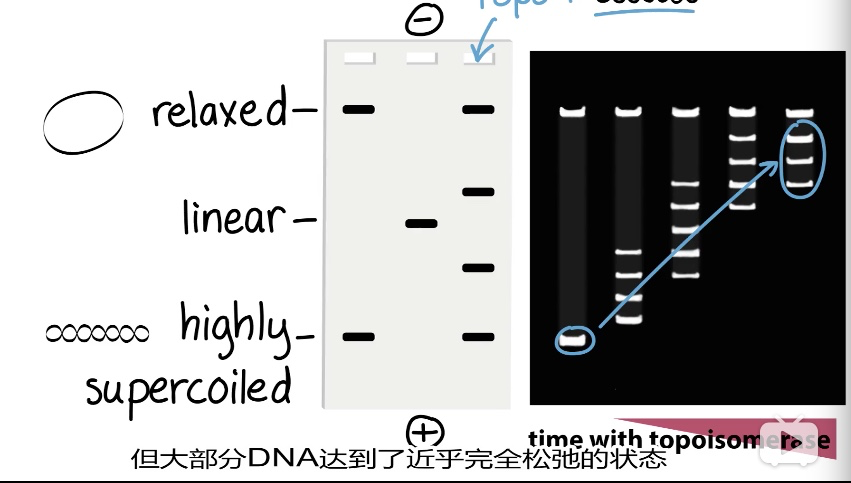
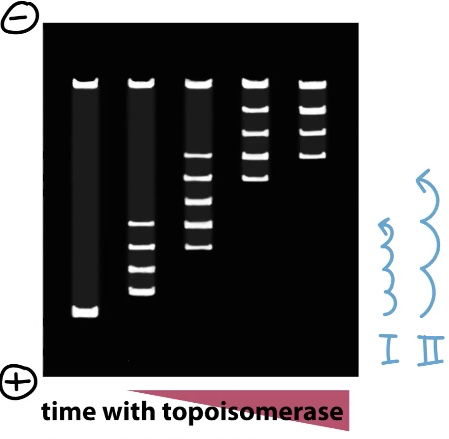
5、Topoisomerase 拓扑酶
DNA是10.4bp每一个螺旋的结构
- type1 :切一个链,不用ATP,依靠DNA链的能量【环绕数一次±1】
- type2:【一次±2】
- 解索烃
- 在细菌中,gyrase让链unwound以利解旋
- 在嗜热菌中,anti-gyrase让链overwound,更稳定

assay for topoisomerase
supercoil 泳动速度快
区分 type1 type2:
- kDNA 连环索烃:短膜虫质粒
看relex的速度 1慢 2快
三、聚合酶的特化 (原核为例)
1、不同的DNA聚合酶
2、滑动夹slide clamp
- 让聚合酶长距离合成而不掉落
- 完成时,由于聚合酶识别到了尽头的双链,构象变化,从滑动夹解除
- 然后滑动夹还会参与其他作用蛋白的募集
3、滑动装载器 装卸滑动夹
- 过程
- ATP作用下,装载器张开, 装载器与滑动夹结合,滑动夹开环
- DNA结合
- ATP水解下, 装载器与滑动夹解离
- 在有TPJ时, 装载器装载滑动夹
- 当滑动夹无用时解离
- 装载器 与 滑动夹的结合蛋白有相同结合位点,故有作用时不会解离
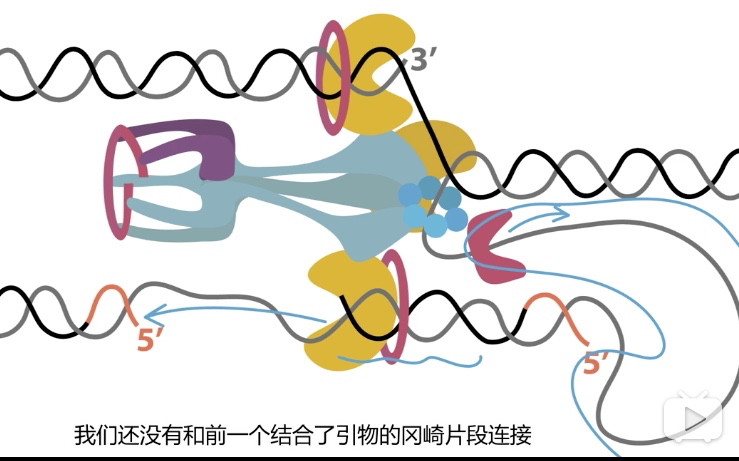
四、复制叉上的合成(原核为例)
长号模型
1、全酶的组成
- 核心酶 x 3
- τ蛋白
- 柔性接头
- 滑动加载器
- 滑动夹
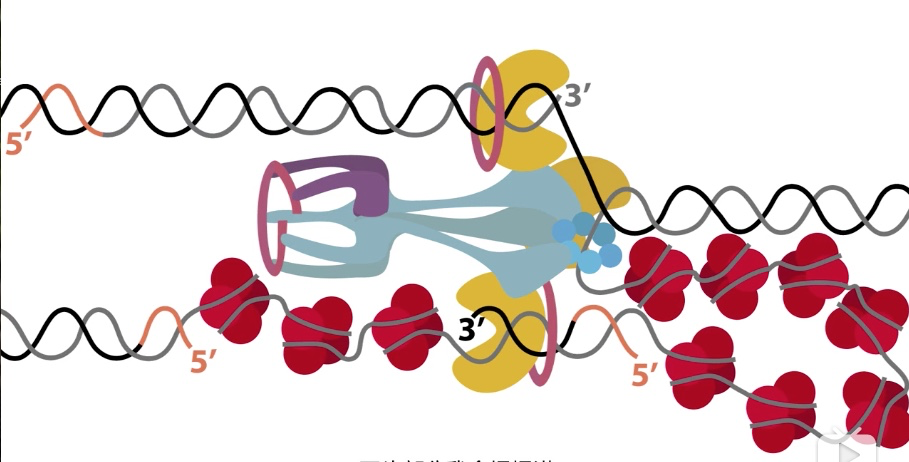
2、复制叉蛋白的相互作用
- 聚合酶通过τ亚基与解旋酶相互作用
- 确保聚合、解旋耦联
- 引物酶与解旋酶相互作用
- 激发引物酶活性,调控冈崎片段长度
HOW THE ENZYMES WORK TOGETHER
1 τ亚基 刺激 解旋酶
2 解旋酶 刺激 引物酶
3 滑动夹 限制聚合酶在DNA上滑动,直到完成
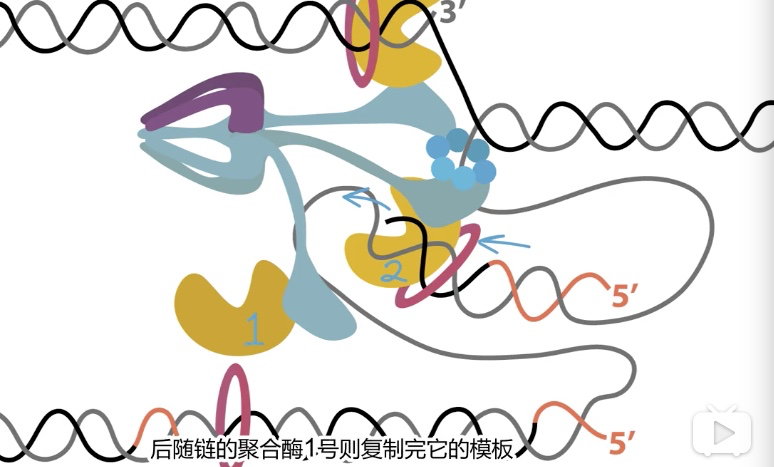
- 后随链解链后,与SSB结合。引物酶被激活后,在后随链上合成引物
- 有两个核心酶参与合成后随链
- 第二个在第一个释放前已经开始合成,释放的聚合酶在τ蛋白的限制下,很快又结合上PTJ,开始新合成
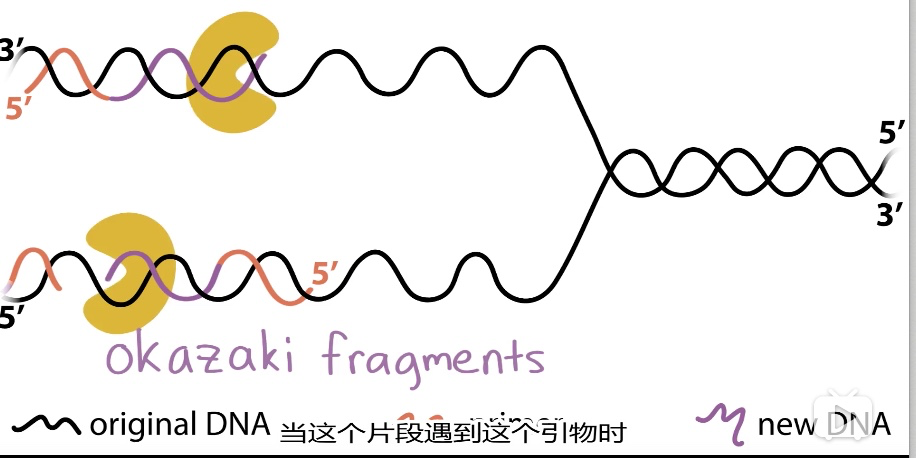
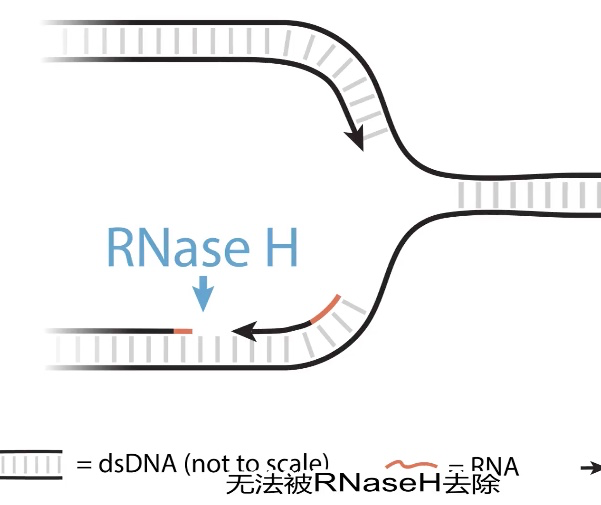
3、冈崎片段的切去修补
- RNase H讲解RNA引物,但不能降解和dNMP结合的那个rNMP
- 再用外切酶降解
- DNA聚合酶填补空隙
- DNA连接酶连接
trombone model of dna rep P293

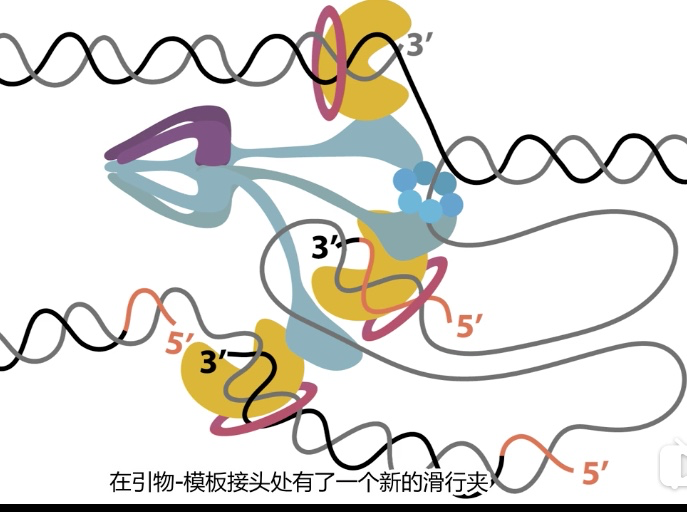
五、复制的起始 (原核为例)
1、特定的基因组DNA序列指导DNA复制的起始
2、复制起始的复制子模型
- replicon : DNA均从称为复制子的特定位置开始复制 控制复制起始的两个原件
- replicator:复制器:指导DNA复制起始的整套DNA顺式作用序列(其中有复制起始点)
- initiator: 起始子:特异识别复制器中一个DNA元件,并且激活复制的起始
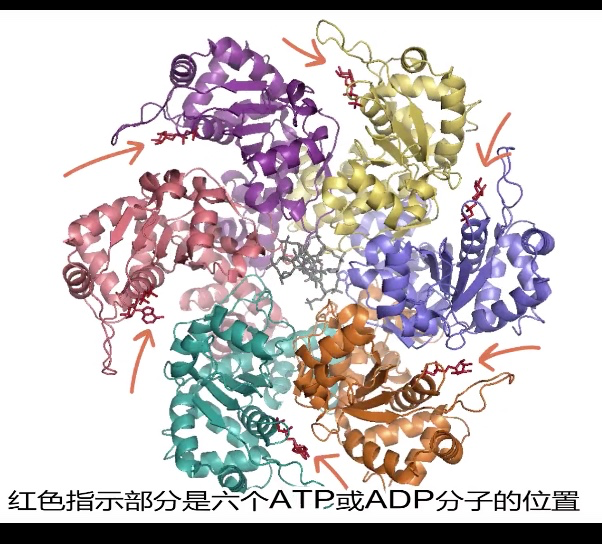
- 依靠ATP的结合与水解调节 核心AAA+ATP基序

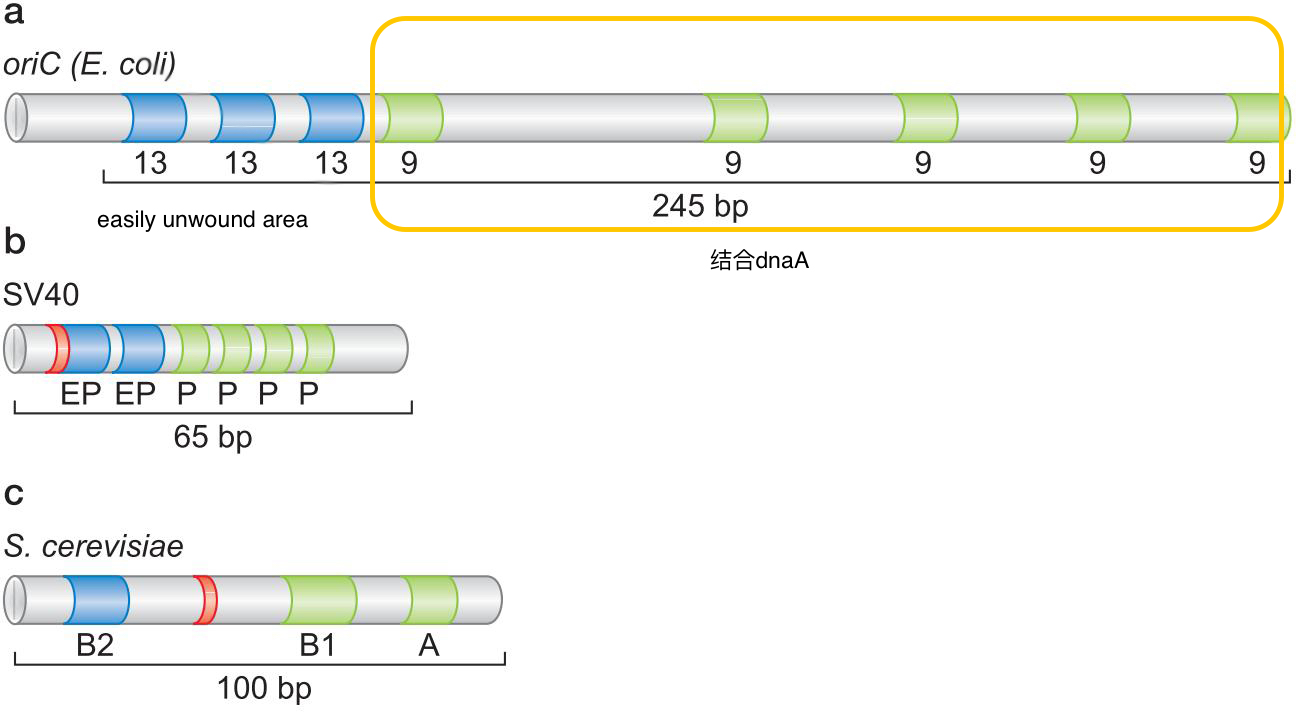
六、结合和解旋 起始子蛋白对复制起始位点的选择和激活(原核为例)
1、蛋白质-蛋白质 与 蛋白质——DNA相互作用 指导起始过程
- 复制机器的组装
- 起始子蛋白结合DNA的特定序列
- Ecoli 中是 dnaA 结合 9mer ,13mer 是easily unwound序列
- 起始子与复制所需的其他蛋白因子相互作用,蛋白因子和蛋白因子相互作用,组装成复制机器
- 起始子蛋白结合DNA的特定序列
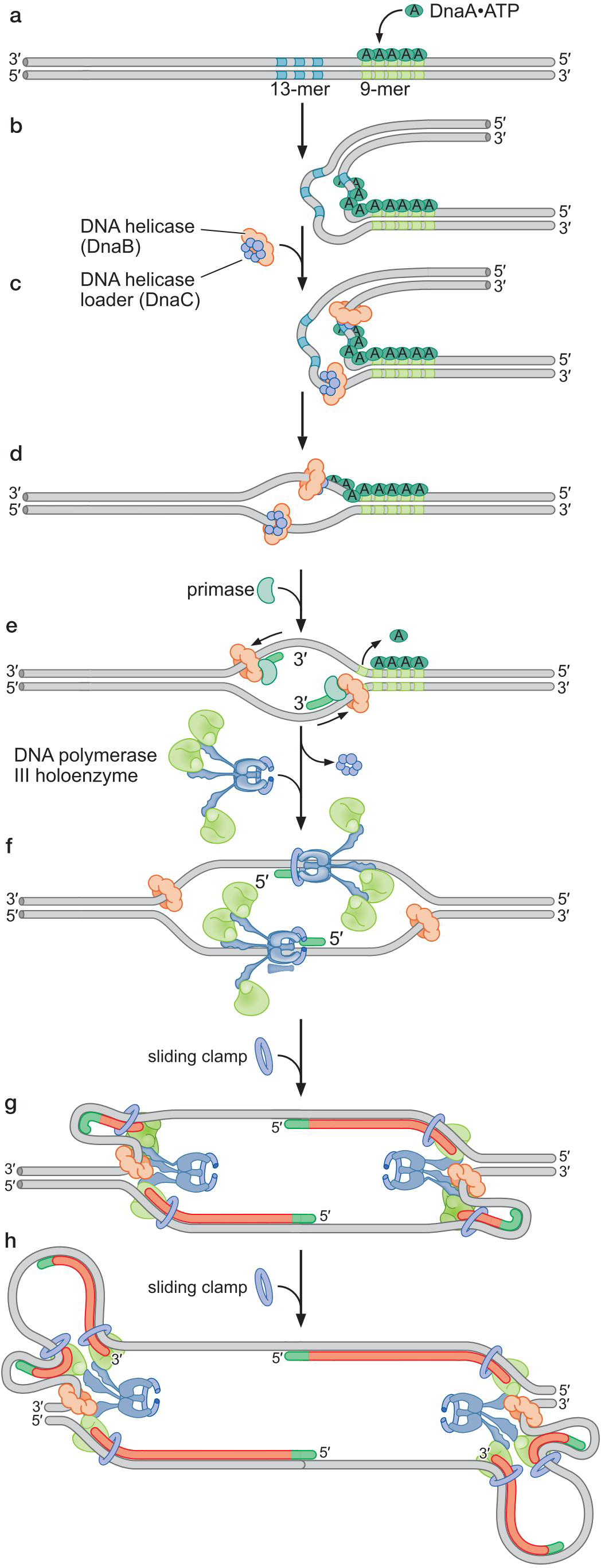
- dnaA结合富含AT的9mer,使得13mer区解链
- 解旋酶在loader的帮助下,结合上dna。因为loader的抑 制,此时解旋酶没有活性
- 解旋酶募集引物酶,引物酶合成引物。引物导致loader解离,解旋酶拥有活性。
- 解旋酶运动,将链上的dnaA除去
- polyIII 全酶的识别解旋酶和引物模板接头,被引导至特定位点。
- 全酶在PTJ处组装滑动夹,滑动夹被聚合酶识别,合成。全酶的另外两个聚合酶位于后随链处
- 解旋酶的作用下,后随链解旋,ssb蛋白包绕
- 解旋酶和全酶τ亚基耦联行动
- 引物酶合成冈崎片段的引物。
- 引物酶受解旋酶刺激,就是,解旋一段,合成一个引物
- 另外两个后随链聚合酶合成冈崎片段
七、真核细胞的复制起始与延续
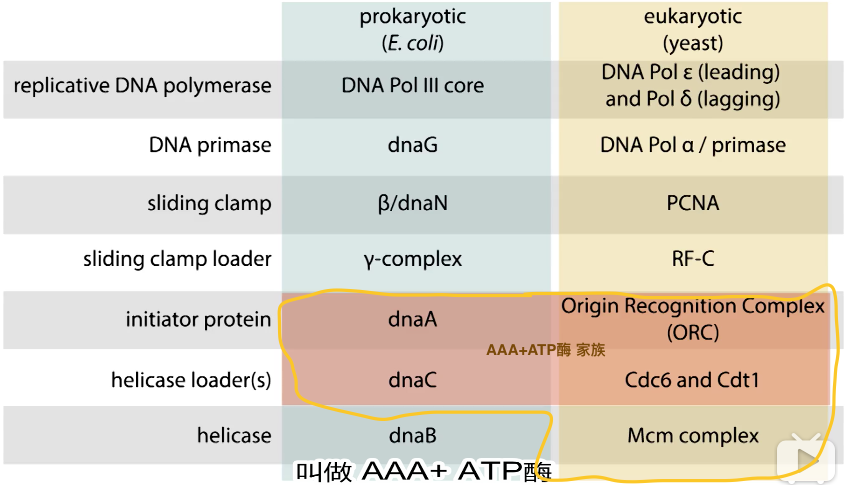
1、真核生物与原核生物酶的比较
2、复制的起始--装载解旋酶
- 复制器上装载解旋酶 G1末期
- ORC募集Cdc6、Cdt1 介导的 装载
- 复制器,起始位点的激活 S 期
- CDK,DDK激活解旋酶
- 两个过程分开,保证了细胞周期中每个染色体复制一次

- 真核起始子识别复制器,结合ATP
- 进入G1期:
- ORC结合起始点(结合ATP)
- Cdc6 结合上ORC(ATP结合)
- Mcm2-7结合Cdt1
- 这相当于解旋酶装载器的两部分分开作用,然后再结合起来
- Cdt1 与 ORC 相互作用,Mcm27——Cdt1 复合体被ORC募集
- Cdc6水解ATP:Mcm2-7头对头二聚体装载
- Mcm2-7包绕双链复制起始点,Cdc6与Cdt1释放
- ORC水解ATP 重新开始一轮循环
- 同一起始位点可以装载多个解旋酶

- 进入S期后,CDK、DDK被激活
- DDK作用于已装载的解旋酶
- CDK作用于Sld2、Sld3
- Sld2、Sld3与Dpb11结合
- 这些蛋白导致解旋酶激活蛋白Cdc45、GINS与解旋酶结合
- 形成CMG复合物
- Cdc45-Mcm2·7-GINS
- Sld2、Sld3解离,Dpb11解离
- Mcm2-7复合体破裂成2,并且各自包绕一条单链
- 三种聚合酶按特定顺序组装
- DNA Pol ε 与Cdc45,GINS同时结合在起点(解旋前)
- DNA Pol δ 与 α(引物酶) 要等解旋后才被募集
- 保证酶结合上了,引物才合成
- 只有 CMG 与 三种聚合酶成为复制机器,继续起作用
- Cdc6 Cdt1 等解离或被破坏
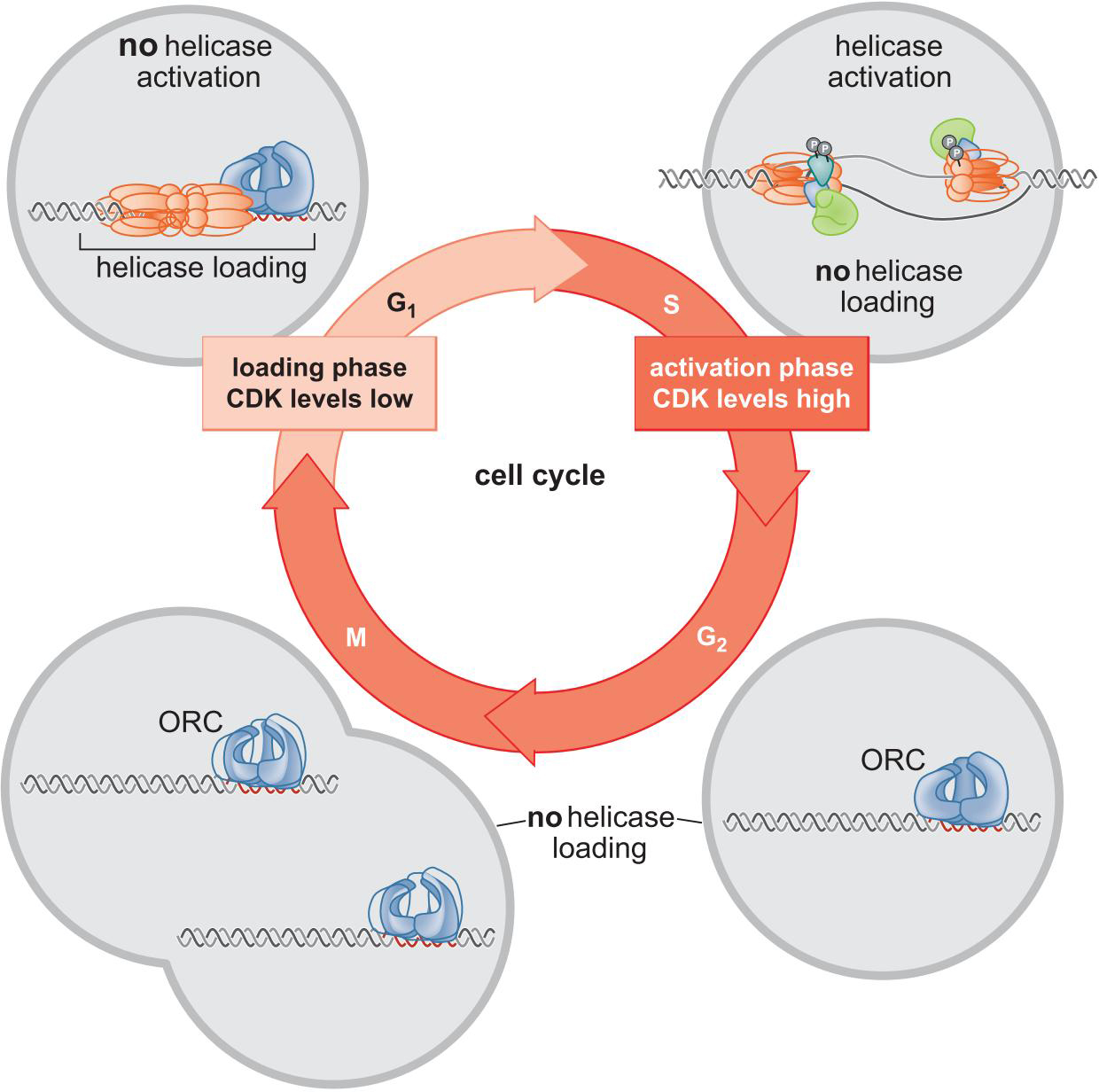
八、每个细胞周期只有一轮复制的发生
真核细胞中有数以百千记的复制起始位点,但是每个细胞周期只能进行一轮细胞复制,否则会导致染色体断裂等损伤。
1)如果一个潜在复制起始位点在相邻的复制机器来临前,并没有开始复制,那么这个位点就会被失活(复制机器把蛋白去除);
2)对解旋酶装载和激活的调控:G1装载,S激活。
调控:与CDK活性有关
- G1期CDK活性低:允许装载,不激活
- S G2 M 期 CDK高,激活,抑制装载
- 在起始点处完成复制后,复制机器离开,会产生一条新链,暴露出复制器,这回让ORC迅速结合,但是CDK水平高,阻止了Cdc6,Cdt1,ORC的相互作用。
九、结束复制
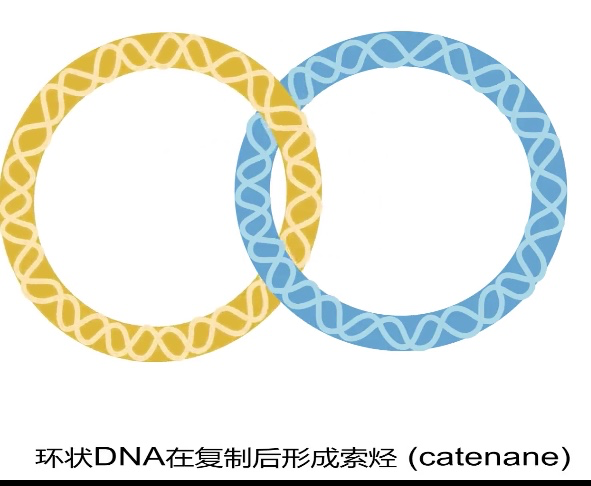
1、子代DNA分子的分离需要拓扑异构酶II
- 环形复制完成如铰链
- 线性性染色体复制完成后有DNA连接成环和蛋白作用,故也有和环形类似的问题
2、后随链的合成不能合成线性染色体的末端(冈崎片段5'端RNA引物切口)
- 细菌的解决方法
- 蛋白质作为末端(通常是酪氨酸提供-OH)
- 真核生物采用端粒来解决:
- 端粒,首尾相接,富含TG的序列(5‘TTAGGG3’)
- 不使用一般聚合酶;使用特殊DNA聚合酶 端粒酶

3、端粒酶
- 组成
- 蛋白亚基
- RNA
- 端粒酶RNA :TER
- 1.5拷贝的序列(人 5'-TAACCCTAA-3')
- 特性:
- 反转录酶活性
- 不需外源模板
- 不能复制RNA模板以外的序列
- 延伸性,核苷酸前体,延长3‘端与普通聚合酶类似
4、端粒学说
- 端粒酶延伸3’末端解决末端复制问题
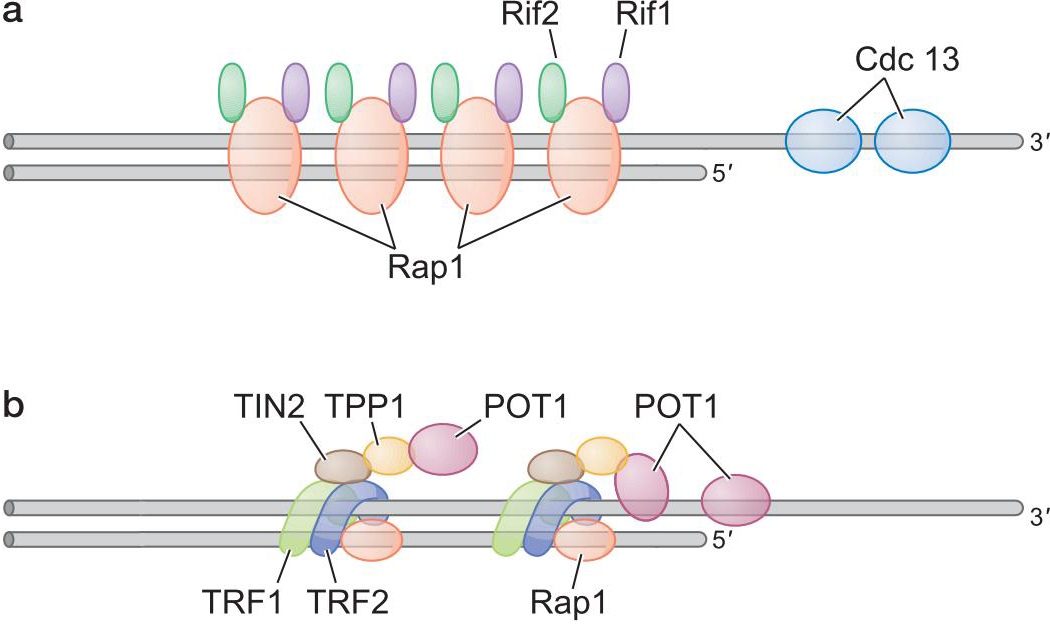
- 端粒结合蛋白调节端粒酶活性 和 端粒长度
- 酵母中
- 端粒结合蛋白Rif 1,2是端粒酶弱抑制剂
- 随着端粒延长,端粒结合蛋白增多,端粒酶活性被抑制
- Cdc 13 是正向激活剂
- 结合端粒ssDNA,募集端粒酶
- 人中
- POT蛋白 抑制作用
- 端粒结合蛋白保护染色体末端
- 断裂标记 ??????
- 结合蛋白
- 形成 t 环
- t环的形成使端粒免受DNA修复酶修复

- 但也能阻止端粒酶的作用
- 端粒越短,越难形成t环
- 这产生对端粒长度的控制
- t环的形成使端粒免受DNA修复酶修复
十、染色体的组装
1、核小体是染色体的结构单位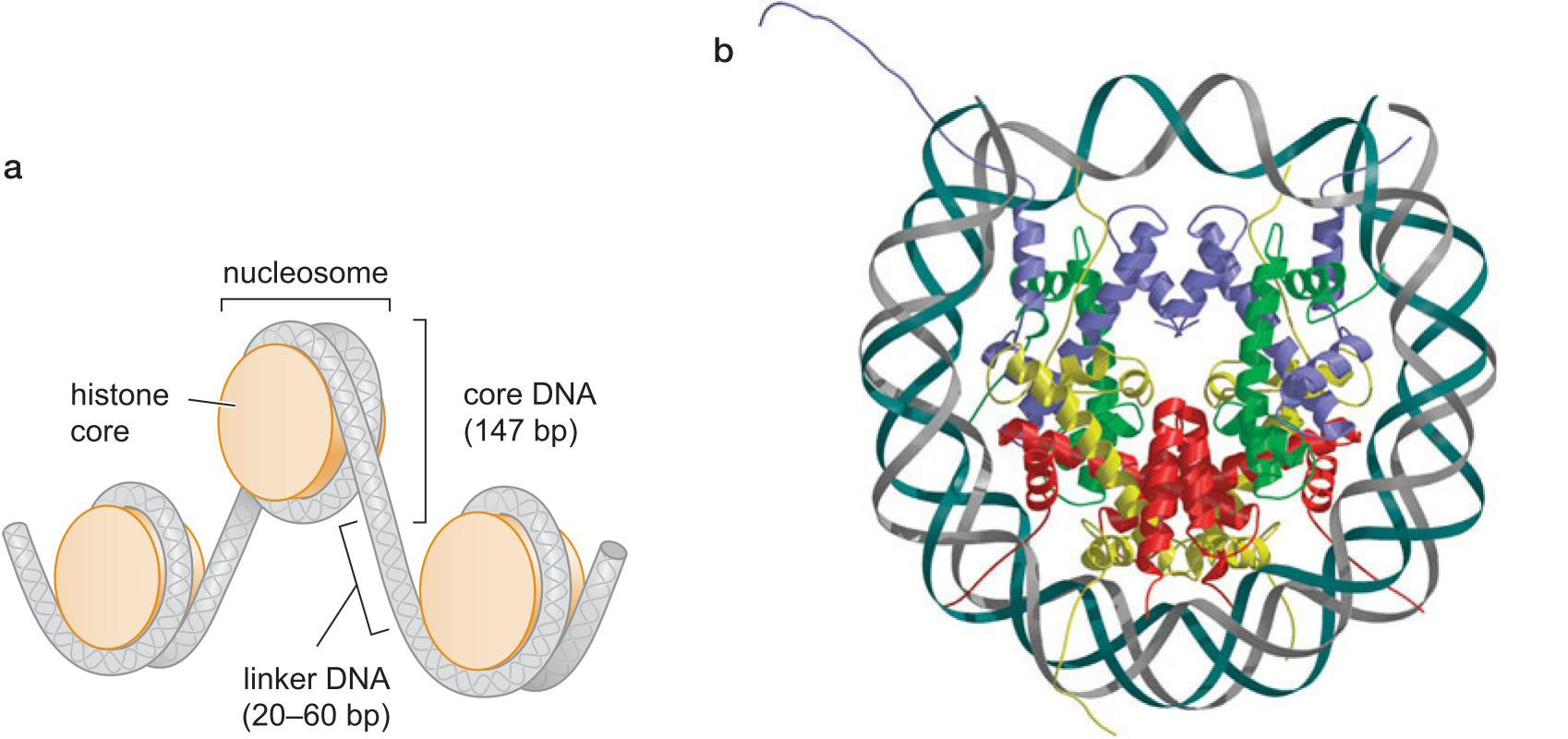
- 8个组蛋白核心
- 核心DNA
- 1.65圈 147bp
- 连接DNA
- 物种差异 20~60bp
2、组蛋白 带正电荷的小分子蛋白质
· 带正电,赖氨酸,精氨酸含量高
连接组蛋白
- H1
核心组蛋白
- H2A H2B
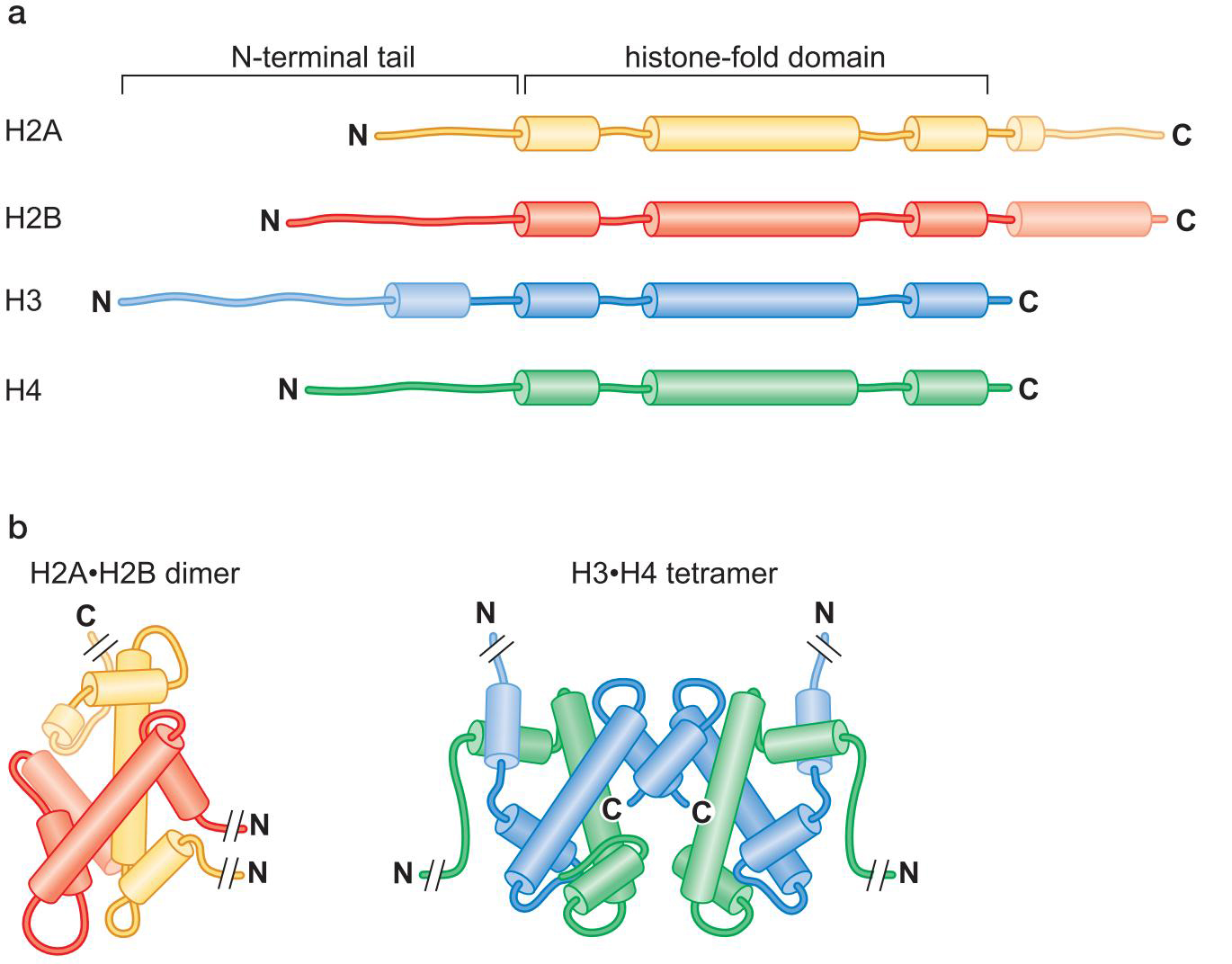
- H3
- H4
每种核心组蛋白都存在一个保守结构域:组蛋白折叠域
组蛋白组装的顺序:
- H3·H4四聚体与 DNA 结合
- 2个 H2A·H2B 二聚体在往上结合
核心组蛋白有 N 端尾巴
3、许多不依赖于DNA序列的接触介导核心组蛋白与DNA的相互作用
- 与小沟和磷酸骨架的作用
- 主要是蛋白质与小沟附件的磷酸骨架氧原子间形成的氢键
- 与碱基也有小作用
- 这些作用DNA弯曲驱动力
4、组蛋白N端尾巴可稳定盘绕在八聚体上的DNA
- 从DNA螺旋之间或两侧伸出尾巴,作用类似于螺丝上的凹槽,指导DNA左手方式盘旋,形成负超螺旋 。
5、盘绕的DNA呈负超螺旋特性
有利于复制、转录
- 真核靠组蛋白: 装配一个核小体 -1.2linking number
- 原核靠gyrase 促旋酶(需ATP)
- 嗜热菌有反促旋酶
6、核小体的组装
- DNA复制后染色体即开始组装
- 复制叉将核小体分开后,两个 H3·H4 二聚体随机去到两条子链上,有助于染色质状态的遗传
- Molecluar Biology of Gene 7th 认为H2A/B 将释放如可溶环境
- 核小体的组装需要组蛋白伴侣(histone chaperone)
- CAF-1的机制
- PCNA是DNA复制时限制聚合酶位置的滑动器 (那个环)
- PCNA与聚合酶解离后,结合组蛋白伴侣
- 优先将H3·H4复合体结合上DNA
- 继续组装步骤 : 2* H2A/B 招募
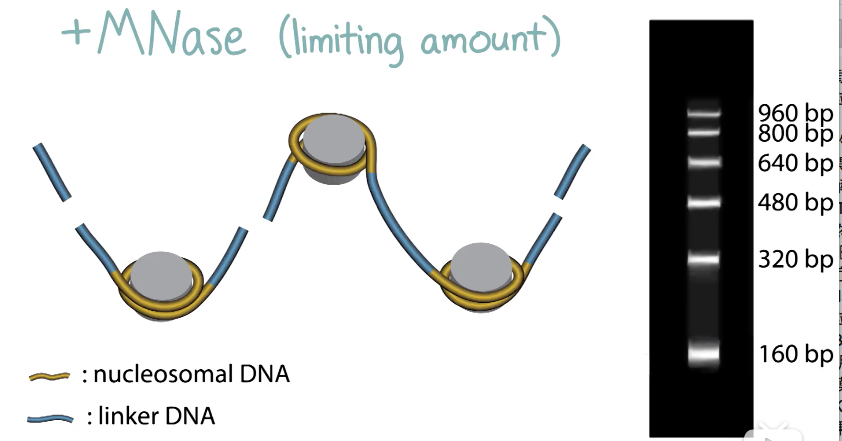
高盐让核小体崩解
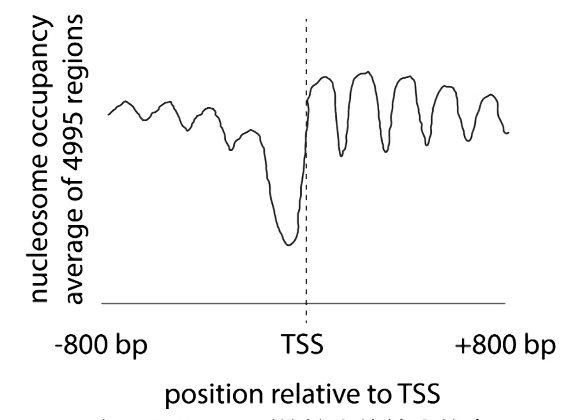
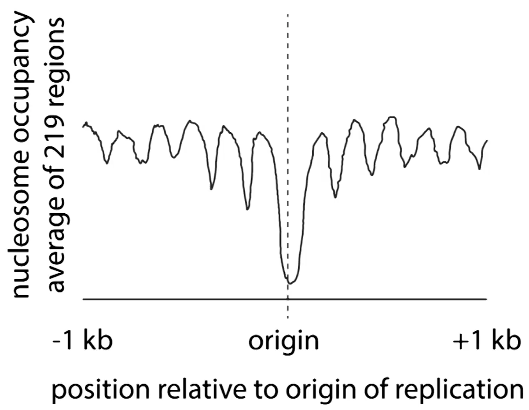
Assay: 定位核小体实验
微球菌核酸酶MNase :双链内切酶,非序列特异
连接dna的长度会有物种差异,但是盘绕核小体的长度都是一样的
Assay: MNase-seq 可以知道位置
1.深度消化 只剩147bp
2.电泳纯化
3.两端深度测序 ??
4.沿染色体定位
细胞周期任何时机都可以做此实验
通过核小体的限制,让复制、转录蛋白结合在正确位置
7、染色体的高级结构
- 异染色质和常染色质
- 组蛋白H1与核小体之间的连接DNA结合
- 核小体束能形成更复杂结构 30nm fiber
- 螺线管模型
- 锯齿模型
-
与连接DNA长度有关
- 进一步凝聚
- 核骨架为中心形成的大环
- 拓扑酶II 保证拓扑学上分离
- SMC 与凝聚、姐妹染色单体联会有关
- 组蛋白变构体影响核小体功能
- 与修复位点识别有关
- 可能干扰动粒结合
- 活性位点裸露在外