目的:本文主要简单介绍pindel检测sv的基本知识
能力:会基本使用,简单结果文件解读
官网:
参考文献:
Ye, K., Schulz, M. H., Long, Q., Apweiler, R. & Ning, Z.
Pindel: a pattern growth approach to detect break points of large deletions and medium sized insertions from paired-end short reads.
Bioinformatics 25, 2865–2871(2009).
pindel变异检测:
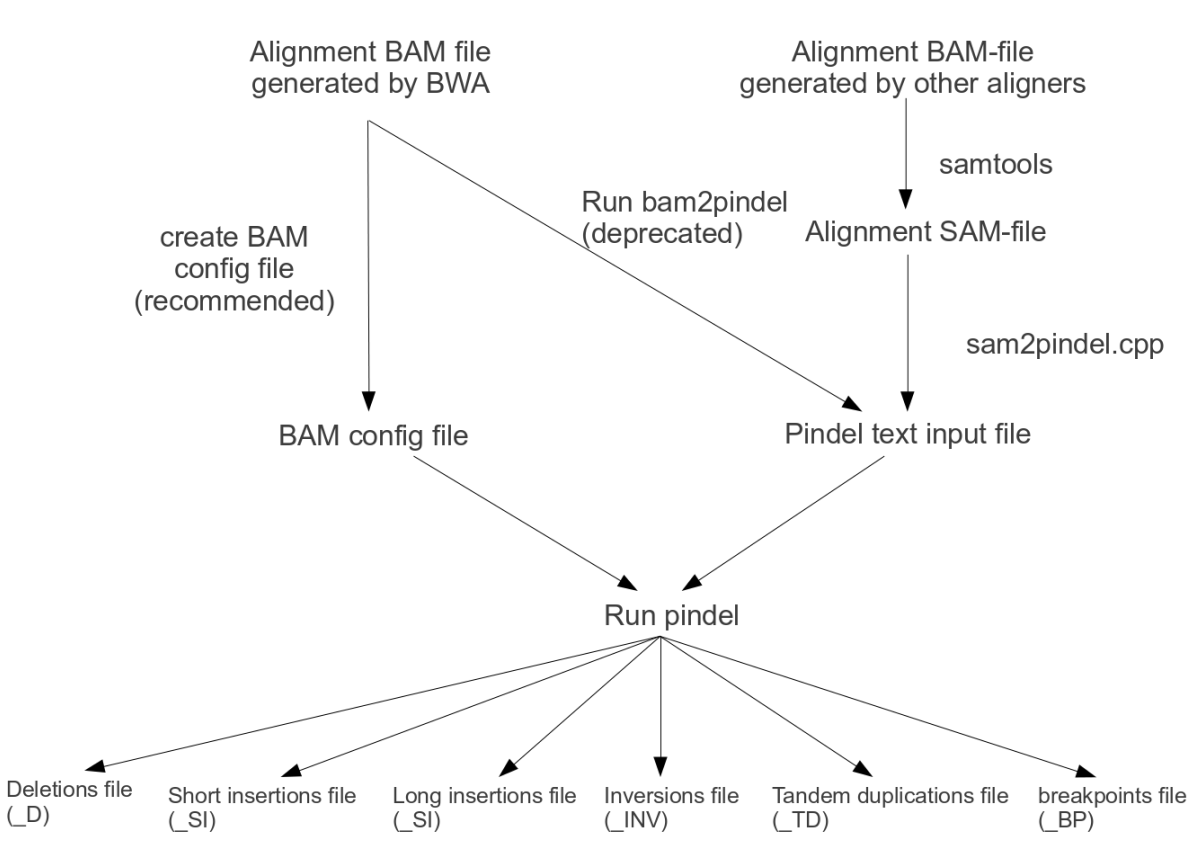
1.pindel进行sv检测时,需要一个配置文件, 配置文件内容如下所示:
$ cat FLT3_28608223_conf edit.sorted.bam 250 FLT3
第一列:bam的绝对路径
第二列:bam的intersize, 写个大概的值即可(本人的测序数据为PE100)
第三列:设一个标签,因为这边可以设多个bam文件,这边的标签就会代替文件名出现在最终的结果中来区分reads的不同来源。列与列之间用制表符或者空格分开。
2.pindel进行sv检测的命令行参数:
/jdfstj1/B2C_COM_P1/pipeline/miniconda3/bin/pindel -f hg19.fa -i FLT3_28608223_conf # 上述描述的配置文件 -c chr13 -o FLT3_28608223
上述命令可以产生多个结果文件: 不同类似的变异结果分开放置.
FLT3_28608223_BP FLT3_28608223_INT_final FLT3_28608223_LI FLT3_28608223_SI FLT3_28608223_TD FLT3_28608223_CloseEndMapped FLT3_28608223_D FLT3_28608223_INV FLT3_28608223_RP
D = deletion 缺失序列
SI = short insertion 短的插入序列
INV = inversion 转位
TD = tandem duplication 串联重复
LI = large insertion 长的插入序列,这个文件的格式跟其他文件的很不相同
BP = unassigned breakpoints 没有分到上面任意一种类型剩下来的断点
3.第二步的结果可能利于我们的阅读,因此可通过以下操作将其转换为vcf文件格式
/jdfstj1/B2C_COM_P1/pipeline/miniconda3/bin/pindel2vcf -r hg19.fa -R hg19 -p FLT3_28608223_TD -d 20201101 # 随便是个啥,没啥用 -v FLT3_28608223_TD.vcf -G#让它尽可能符合GATK输入文件的要求。