现有数据维度:
PassengerId
survival 生存 0 = No, 1 = Yes
pclass 票类 社会经济地位,1 = Upper, 2 = Middle, 3 = Lower
sex 性别
Age 年龄
sibsp 兄弟姐妹/配偶在泰坦尼克号上
parch 父母/孩子在泰坦尼克号上
ticket 票号
fare 客运票价
cabin 舱位数量
embarked 始发港 C = Cherbourg, Q = Queenstown, S = Southampton
读取数据,可知船上共有891名乘客,其中男性577人,女性314人,乘客生还率为38.38%,有年龄信息的乘客平均年龄为29岁,且数据需进行以下预处理:
1、年龄存在缺失,进行中位数填补
2、姓名这一列中间值也许有用,进行提取
3、cabin存在较多的缺失
4、登录港口存在较少缺失,使用明显最多的登录港口进行填充
5、船票开头的标记本例中不进行处理分析
> titanic<-read.csv('titanic.csv')
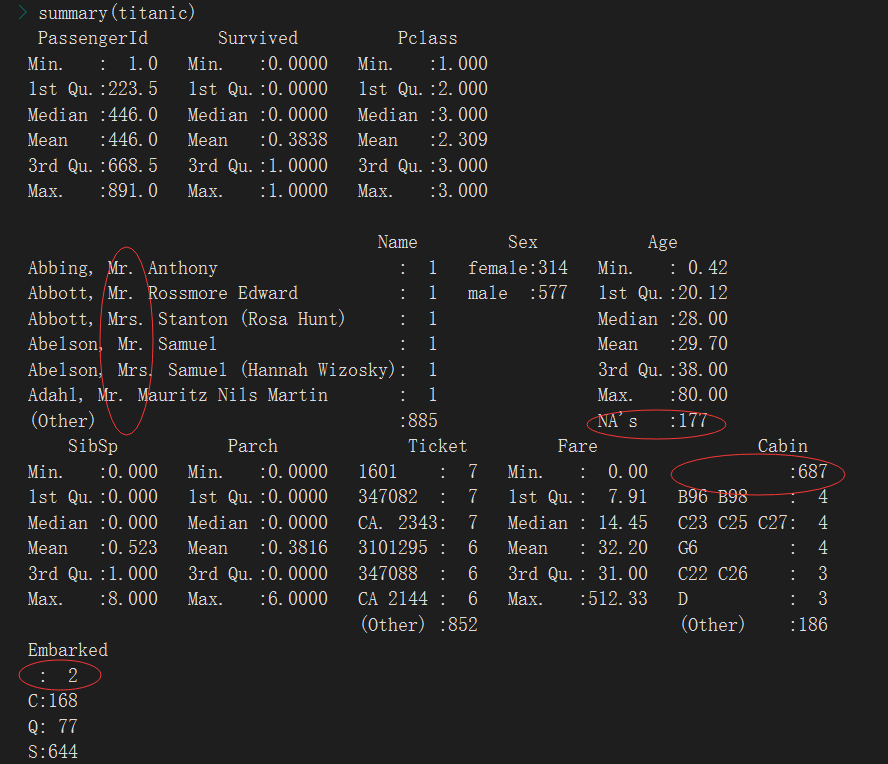
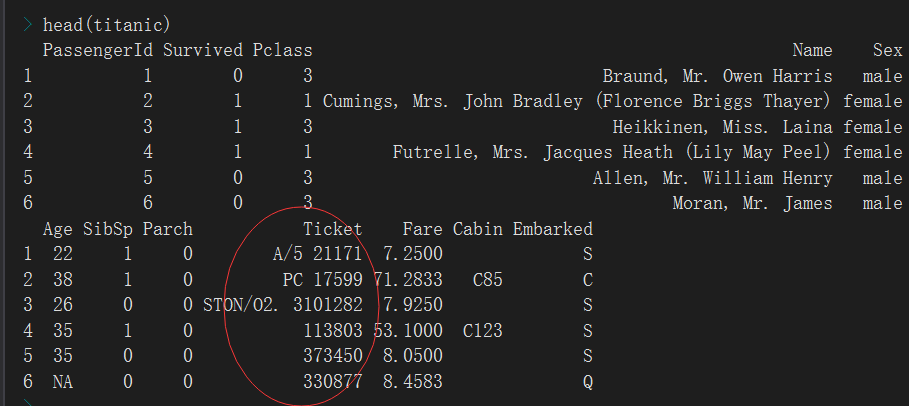
pclass应该是分类变量,是否幸存也是分类变量但是后续需要使用暂时不做变化,只把Pclass转换为因子,确实是
> titanic$Pclass <- factor(titanic$Pclass)

中位数填补年龄缺失,登录港口缺失填补为S
median(titanic$Age,na.rm = T) titanic$Age[is.na(titanic$Age)] <- median(titanic$Age,na.rm = T) titanic$Embarked[is.na(titanic$Embarked)] <- 'S'
提取姓名中间的称谓
#首先需要自定义一个函数截取出来称谓
> nametitle <- function(x) { + temp <- unlist(strsplit(x, ','))[2]#strsplit结果为list,要提取第二列转换为unlist + temp <- unlist(strsplit(temp, ' '))[2]#姓名称谓之前存在一个空格,空格之后为我们所需要的称谓 + return(temp)}
#将姓名列转换为字符型向量
titanic$Name <- as.character(titanic$Name)
#测试一下
> nametitle(titanic$Name[1])
[1] "Mr."
#对整列应用提取函数,并赋值给新的一列名为title
> titanic$title <- unlist(lapply(titanic$Name, nametitle))
#未知称谓标记为未知
> titanic$title[!titanic$title %in% c('Miss.', 'Mr.', 'Mrs.', 'Ms.')] <- 'unknow'
#转换为因子变量
> titanic$title <- factor(titanic$title)
cabin处理,由结果可知大多数人其实都没有关联舱位,或者说可能有但是关联舱位的数据缺失严重
> CabinNum <- function(x) { + if (is.na(x)) { + return(0) + } else { + return(length(unlist(strsplit(x, ' ')))) + } + } > titanic$Cabin <- as.character(titanic$Cabin) > titanic$CabinNum <- unlist(lapply(titanic$Cabin, CabinNum))
name、cabin、票号变量基本上可以去除
> titanic$Name <- NULL > titanic$Cabin <- NULL > titanic$Ticket <- NULL
目前的数据已经没缺失,接下来针对分类变量进行哑变量处理
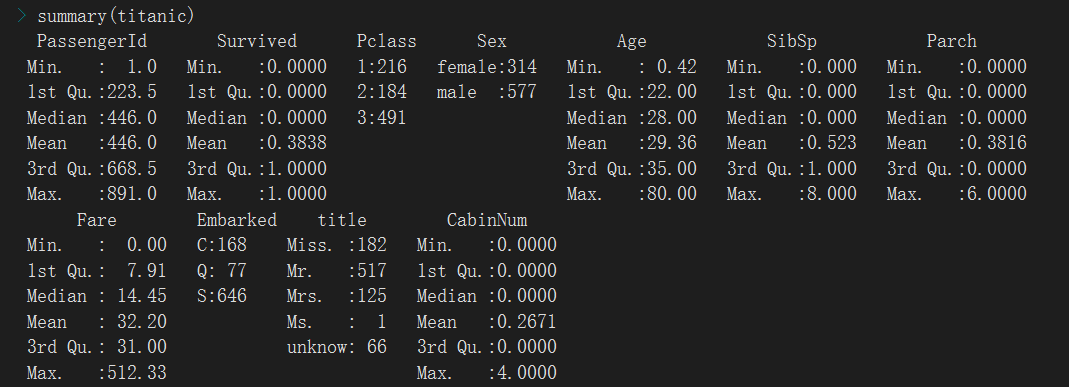
> base <- data.frame(predict(dummyVars(~., data = titanic), titanic)) > describe(base)

将数据集进行切割,切分为训练集和测试集
> trainid <- createDataPartition(base$PassengerId, p = 0.75, list = F) > train <- base[trainid,] > test <- base[-trainid,]

方法:逻辑回归
初次建模
> logistic <- glm(Survived ~ ., + data = train[, -1], + family = 'binomial'(link = 'logit')) + + summary(logistic)

选择sig较高的值再次进行建模
> logistic <- glm(Survived ~ Pclass.1 + + Pclass.2 + + Age + + SibSp + + title.Mr., + data = train[, -1], + family = 'binomial'(link = 'logit')) + + summary(logistic)
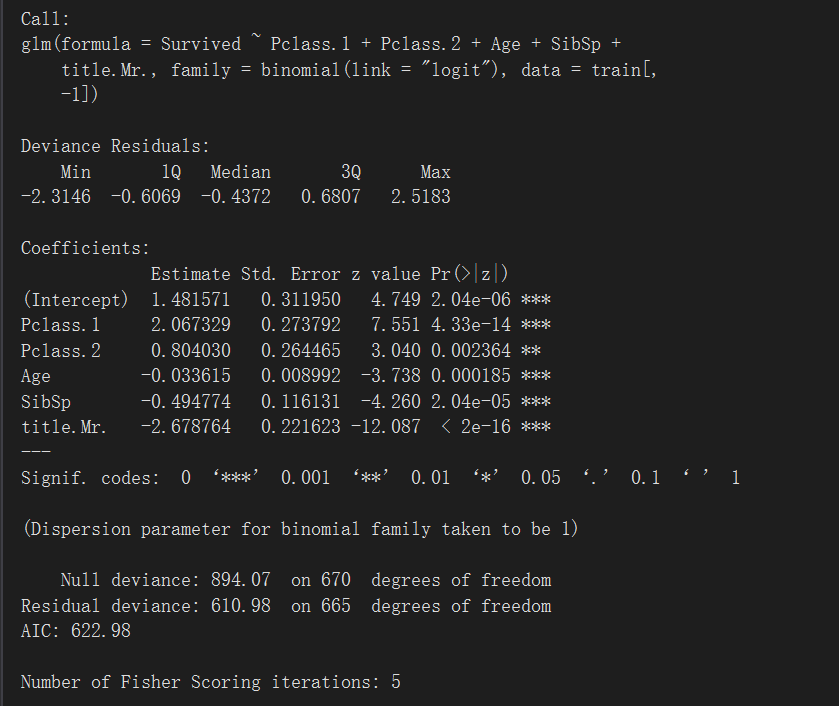
用之前生成的测试集对模型进行评估
> test$predict <- predict(logistic, test, type = 'response') + test$predictClass <- NULL + test$predictClass[test$predict >= 0.5] <- 1 #逻辑回归得到的是一个概率值,而如何去划分需要我们指定,本例中大于等于0.5为正例,即:幸存。0.5也可以划分为0,已经测试过这个例子中不影响预测结 + test$predictClass[test$predict < 0.5] <- 0 + table(test$Survived, test$predictClass)

> table(test$Survived) 0 1 136 84
由以上结果可知:测试集内的对象而言,136个0预测正确的有119个,84个1预测正确的有66个,模型效果尚可。
可以再调整一下,我们知道女士更容易获救,而本次建模中female变量没有进去(将称谓中的mr变量换成female),再试试
> logistic <- glm(Survived ~ Pclass.1 + Pclass.2 + Sex.female + SibSp,data = train[,-1],family = 'binomial'(link='logit')) > summary(logistic)

效果没有原先的好,在考虑共线性的基础上,可以自己尝试叠加不同变量,检验模型效果,可同时结合建模目的进行变量挑选。
方法二:k近邻
> train <- base[trainid,] > test <- base[- trainid,]
定义标准化函数并且进行标准化#此里中未处理异常值
> normalize <- function(x) { + return((x - min(x)) / (max(x) - min(x))) + } + train$Age <- normalize(train$Age) + train$CabinNum <- normalize(train$CabinNum) + train$SibSp <- normalize(train$SibSp) + train$Parch <- normalize(train$Parch) + train$Fare <- normalize(train$Fare) + test$Age <- normalize(test$Age) + test$CabinNum <- normalize(test$CabinNum) + test$SibSp <- normalize(test$SibSp) + test$Parch <- normalize(test$Parch) + test$Fare <- normalize(test$Fare)
> library(class) > knn(train[,-c(1,2)],test[,-c(1,2)],train$Survived,k = 1) [1] 1 0 1 0 1 0 0 1 1 0 0 0 1 0 0 0 0 1 0 0 0 0 1 1 1 0 0 1 1 0 1 0 0 1 0 1 1 0 0 1 1 0 0 1 1 1 1 0 0 1 [51] 0 0 0 0 0 0 0 0 1 0 0 0 0 1 0 1 1 1 0 0 1 1 1 1 0 1 1 1 1 1 1 1 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 1 0 [101] 0 0 0 1 0 1 0 0 0 0 1 1 1 0 0 0 0 1 0 0 0 0 0 0 1 0 0 1 1 0 1 0 1 1 1 0 0 1 1 0 1 0 0 0 0 1 0 0 0 1 [151] 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 1 0 0 1 1 1 0 0 0 0 0 1 1 1 0 0 0 0 1 0 1 0 1 0 0 0 1 0 0 0 0 0 [201] 0 0 1 0 0 1 1 1 0 0 0 0 0 1 1 1 1 0 0 1 Levels: 0 1 > table(test$Survived, knn(train[, - c(1, 2)], test[, - c(1, 2)], train$Survived, k = 1)) 0 1 0 114 22 1 25 59
可以通过调节K的数量来查看模型效果
> table(test$Survived, knn(train[, - c(1, 2)], test[, - c(1, 2)], train$Survived, k = 3)) 0 1 0 122 14 1 24 60 > table(test$Survived, knn(train[, - c(1, 2)], test[, - c(1, 2)], train$Survived, k = 5)) 0 1 0 124 12 1 21 63 > table(test$Survived, knn(train[, - c(1, 2)], test[, - c(1, 2)], train$Survived, k = 7)) 0 1 0 126 10 1 19 65 > table(test$Survived, knn(train[, - c(1, 2)], test[, - c(1, 2)], train$Survived, k = 9)) 0 1 0 127 9 1 22 62
K=7时,学得最好,不过考虑到运算开销,可以选择K=3作为模型,因为效果差异不太大。
K为奇数,因为要投票
方法三:朴素贝叶斯,只能作用在分类变量上。使用哑变量处理之前的数据,选择其中的分类变量
>install.packages(e1071) > library(e1071)
> trainid<-createDataPartition(titanic$PassengerId,p = 0.75,list = F)
> train<-titanic[trainid,]
> test<-titanic[-trainid,]
> train$Survived <- factor(train$Survived)
+ test$Survived <- factor(test$Survived)
> fit <- naiveBayes(train[, c('Pclass', 'Sex', 'Embarked', 'title')], train$Survived) > table(test$Survived, predict(fit, test)) 0 1 0 121 22 1 23 54
#用laplace修正结果,本例中自变量少,修正后对结果无影响,对于多自变量的模型效果较为明显
> fit <- naiveBayes(train[, c('Pclass', 'Sex', 'Embarked', 'title')], + train$Survived, + laplace = 5) + + table(test$Survived, predict(fit, test)) 0 1 0 121 22 1 23 54
方法四:决策树,既能做回归,也能做分类,这是分类‘class’
trainid <- createDataPartition(titanic$PassengerId,p = 0.75,list = F) train <- titanic[trainid,] test <- titanic[- trainid,] fit <- rpart(Survived ~ ., data = train[,-1], method = 'class') > fit n= 671 node), split, n, loss, yval, (yprob) * denotes terminal node 1) root 671 265 0 (0.60506706 0.39493294) 2) title=Mr. 386 64 0 (0.83419689 0.16580311) * 3) title=Miss.,Mrs.,unknow 285 84 1 (0.29473684 0.70526316) 6) Pclass=3 129 64 0 (0.50387597 0.49612403) 12) Fare>=24.80835 28 2 0 (0.92857143 0.07142857) * 13) Fare< 24.80835 101 39 1 (0.38613861 0.61386139) 26) Fare>=13.90835 37 18 0 (0.51351351 0.48648649) 52) Fare< 14.8729 8 0 0 (1.00000000 0.00000000) * 53) Fare>=14.8729 29 11 1 (0.37931034 0.62068966) * 27) Fare< 13.90835 64 20 1 (0.31250000 0.68750000) * 7) Pclass=1,2 156 19 1 (0.12179487 0.87820513) 14) Sex=male 26 13 0 (0.50000000 0.50000000) 28) Age>=17 16 3 0 (0.81250000 0.18750000) * 29) Age< 17 10 0 1 (0.00000000 1.00000000) * 15) Sex=female 130 6 1 (0.04615385 0.95384615) * > fancyRpartPlot(fit)#需要用到四个包:rattle

> table(test$Survived, predict(fit, test, type = 'class')) 0 1 0 127 16 1 21 56
模型评估的另一种方式:
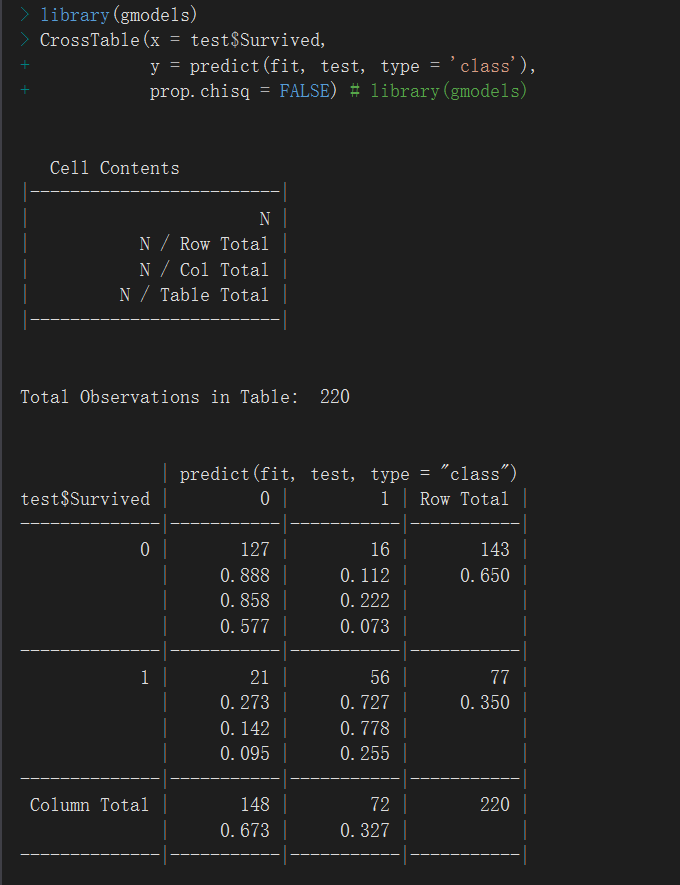
混淆矩阵相关知识:
预测准确率=正确预测的正反例数 / 总数
误分类率 = 错误预测的正反例数 / 总数
真阳性率(召回率):正确预测到的正例的数量/实际上正例的个数,在实际应用中覆盖率是很重要的指标,
阳性预测值=正确预测到的正例数 / 预测正例总数
真阴性率 = 正确预测到的负例个数 / 实际负例总数
阴性预测值 = 正确预测到的负例个数 / 预测负例总数