Wang H, Xu X, Vieira F G, et al. The power of inbreeding: NGS-based GWAS of rice reveals convergent evolution during rice domestication[J]. Molecular Plant, 2016, 9(7): 975-985.
本研究中,作者以1.5x的平均深度,对来自United States Department of Agriculture Rice Mini-Core Collection的203个表型特征鲜明的水稻品种进行了测序,并针对直链淀粉含量、种子长度和果皮颜色三个性状进行了GWAS分析。
在背景介绍中,作者提到,人类研究中即使测序深度极低(0.1-0.5x),基于NGS的GWAS也有着与基于SNP array的GWAS相当的统计分析能力,但这有赖于一个全面的reference haplotypes panel。在水稻中,GWAS的数据既来自于SNP array,又来自于sequencing-based genotyping strategies等,同时基于genome sequencing策略的GWAS也已在谷子、高粱等农作物中得到广泛应用。然而,尚无有关测序深度对近交基因组农作物GWAS定位能力影响的研究。因此,本研究中探索了无reference panel下,低测序深度对近交种GWAS定位能力的影响。由于采用了近交种,作者表示,近交种的基因组是“有效的单倍体”(effectively haploid),从而避免了二倍体数据中genotype calling时的问题。
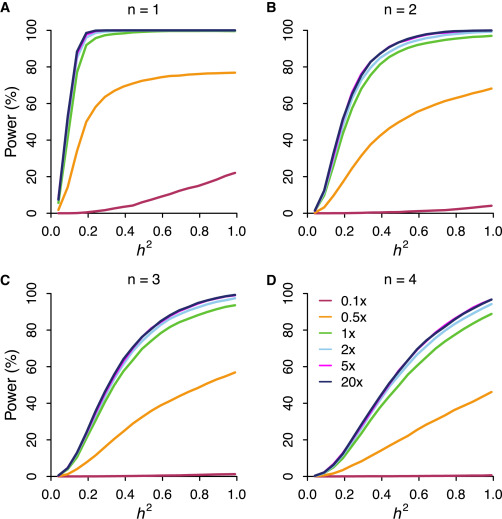
理论模拟测序深度对近交种GWAS的影响
通过模拟,作者得出结论认为对于近交物种GWAS中,测序深度建议值为1-2x。下图中n代表对性状起调控的locus数量(作者提到“... for a Mendelian locus (n = 1)...”),不同颜色的线代表不同测序深度(模拟数据),纵坐标代表mapping power, 横坐标代表(h^2)(narrow-sense heritability of a trait)。
实际测序并imputation
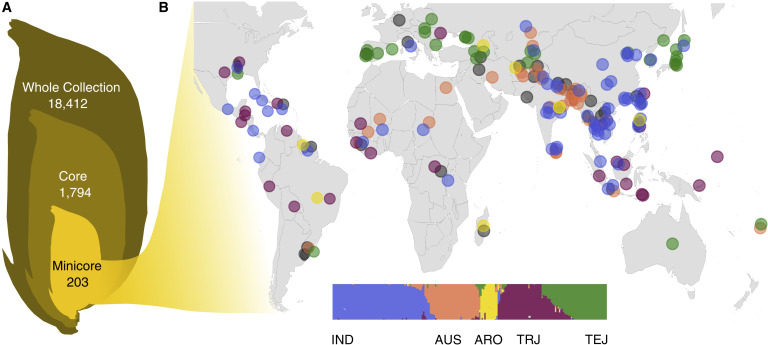
下图展示了水稻Collection的层级结构。作者采用了Mini-Core Collection中的203个样本进行了测序,样本来自75个国家,跨越了“基因多样性与种植地区”,共产生了1280 million个读段,平均每个accession的测序深度为1.5x。总体上,每个accession的65%的基因组区域都有被至少一条高质量读段所覆盖。通过使用ANGSD,作者获取了2.3 million的高可信度SNP,相当于每千碱基6个SNP。
驯化的稻米大部分是自花授粉植物,稻米种质通过自交在种子库中保存了许多代。因此,预期它们的基因组大部分是纯合的。根据这一期望,我们发现大多数水稻品种的近交系数(F)估计值都接近于1。基于这一F值,作者通过SNP推断了genotype[1],并得到了总体缺失率32.8%的genotype数据集。作者通过Sanger测序评估了基因型数据集的准确性。从两个实验获得的基因型之间的一致性率为98.9%。推算(imputation)后,可以完全推断出丢失的数据,一致性比率保持在98.5%(补充表2)。
GWAS
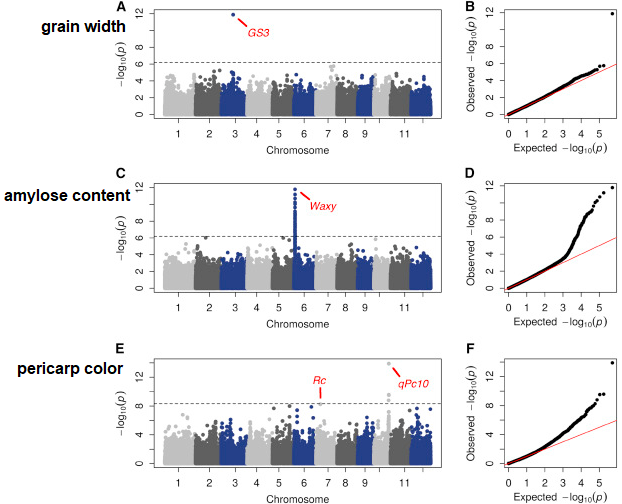
随后,作者对直链淀粉含量、种子长度和果皮颜色三个性状进行了GWAS,并在aus(澳大利亚)群体中发现了一个控制果皮颜色的新locus。下图为GWAS结果(有改动)。
相关参考文献
Vieira, Filipe G., et al. "Estimating inbreeding coefficients from NGS data: impact on genotype calling and allele frequency estimation." Genome research 23.11 (2013): 1852-1861. ↩︎