本周最新文献速递20211031
一、精细解读文献 一
文献题目: An open approach to systematically prioritize causal variants and genes at all published human GWAS trait-associated loci
不想看英文题目: 在所有已发表的人类 GWAS 性状相关位点上系统性优先排序因果变异位点和基因
杂志和影响因子: Nat Genet (IF: 38.33; Q1)
研究意义: 全基因组关联研究 (GWAS) 发现了许多与复杂性状相关的变异位点,但确定因果基因仍是一项重大挑战。本研究提供了一个开放性资源,可在 133,441 个已发表的人类 GWAS 基因座中提供系统的精细定位和基因优先排序结果。
结论:
- 作者从 GWAS Catalog 和 the UK Biobank 总共收集了 3,621 个 GWAS 数据集,133,441 个表型相关位点;
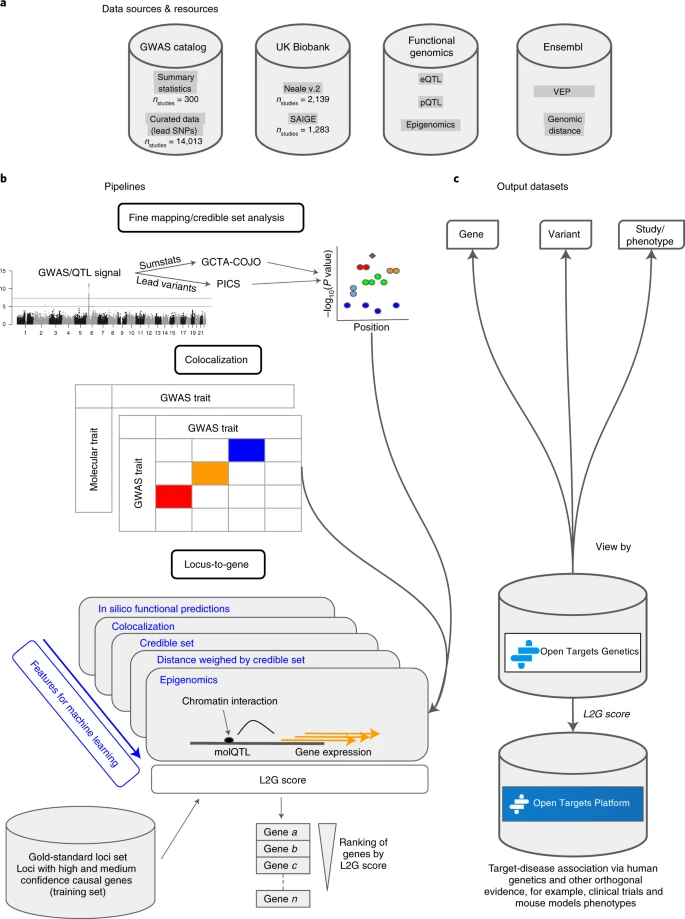
- 133,441 个表型相关位点中,53% 的位点在一种以上的表型间共享;
- 对 133,441 个表型相关位点构建 95% 置信度数据集, 12,500 (9%) 的 95% 置信度数据集只有一个因果位点,21,279 (16%) 的 95% 置信度数据集有 2-5 个因果位点。因果变异位点数量越少的 95% 置信度数据集,通常频率也越低;
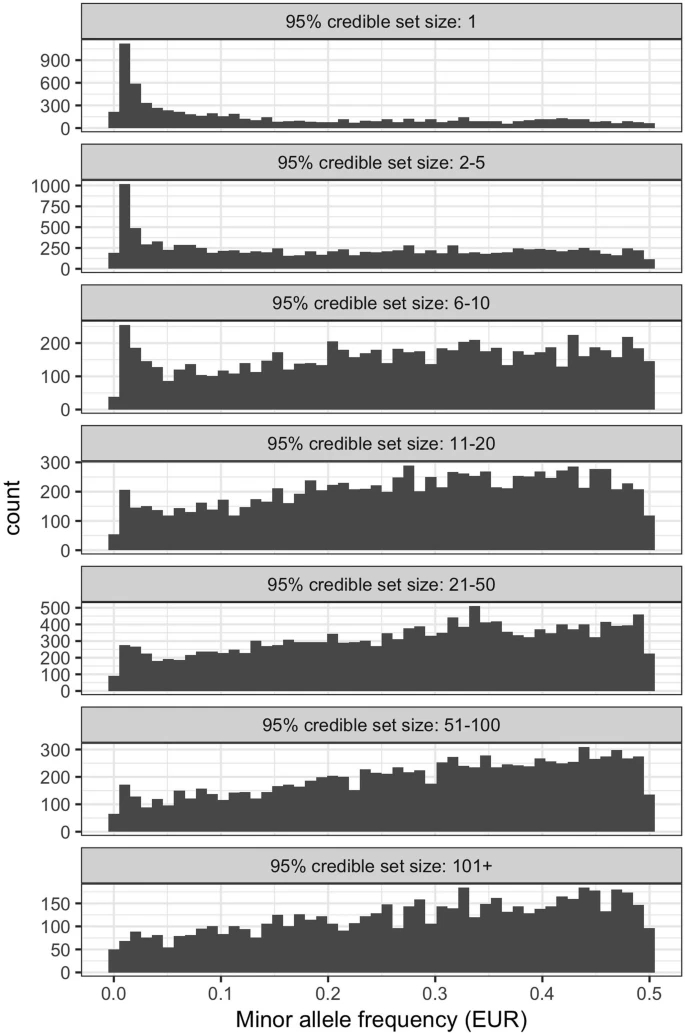
- VEP注释结果表明,与拥有多个变异位点的 95% 置信度数据集相比,只有单个变异位点的 95% 置信度数据集,变异位点对转录本的影响更大 (odds ratio (OR) = 8.51, P < 2.2 × 10 -16 , Fisher’s exact test);
- 保留 95% 置信度数据集中具有中/高影响的 2,284 个编码变异位点(VEP注释),与之相关的编码基因和表型有 378 和 303 个。其中有几个疾病相关的编码基因已被批准为治疗靶点,以 PHLDA3 上的 rs35383942 位点为例, rs35383942 为乳腺癌相关的位点,_PHLDA3_是 TP53 的直接靶标;
- 保留 95% 置信度数据集中具有中/高影响的 2,284 个编码变异位点(VEP注释),与之相关的编码基因和表型有 378 和 303 个。其中有几个疾病相关的编码基因已被批准为药物治疗靶点,以 PHLDA3 上的 rs35383942 位点为例, rs35383942 与乳腺癌相关,而 PHLDA3 被报道为 TP53 的直接靶标,其通过抑制 AKT1 产生抗肿瘤作用;
- 对 70,364 个基因座与表达相关变异位点(eQTL)、蛋白质水平相关变异位点(pQTL)进行共定位, 发现 49.4% 的基因座没有共定位基因( H4 >0.8),25.5% 有一个共定位基因,25.2% 超过一个共定位基因;
- 对 3,621 个 GWAS 数据集进行了跨性状共定位,以识别潜在的受相同分子调控的性状,跨性状共定位揭示 6 号染色体上的一个基因座与哮喘 (6_90220794_T_C) 和克罗恩病 (6_90263440_C_A) 共定位,表明这两种疾病可能在该基因座具有相同的遗传风险;
- 随后作者将精细定位与功能基因组学特征相结合,开发了“基因座-基因”( locus to gene,L2G)模型。在该模型中手动纳入 445 个基因作为金标准(GSP),利用多个功能基因组学特征(比如药物靶点-疾病信息、共定位表观遗传标记、报告基因检测等)进行训练,通过预测 445 个基因是否为因果基因评估模型的表现性能。利用 L2G 模型对所有 GWAS 中的因果基因进行优先排序,发现 L2G 模型鉴定的因果基因被大多数文献支持且富集了更多的药物靶点;
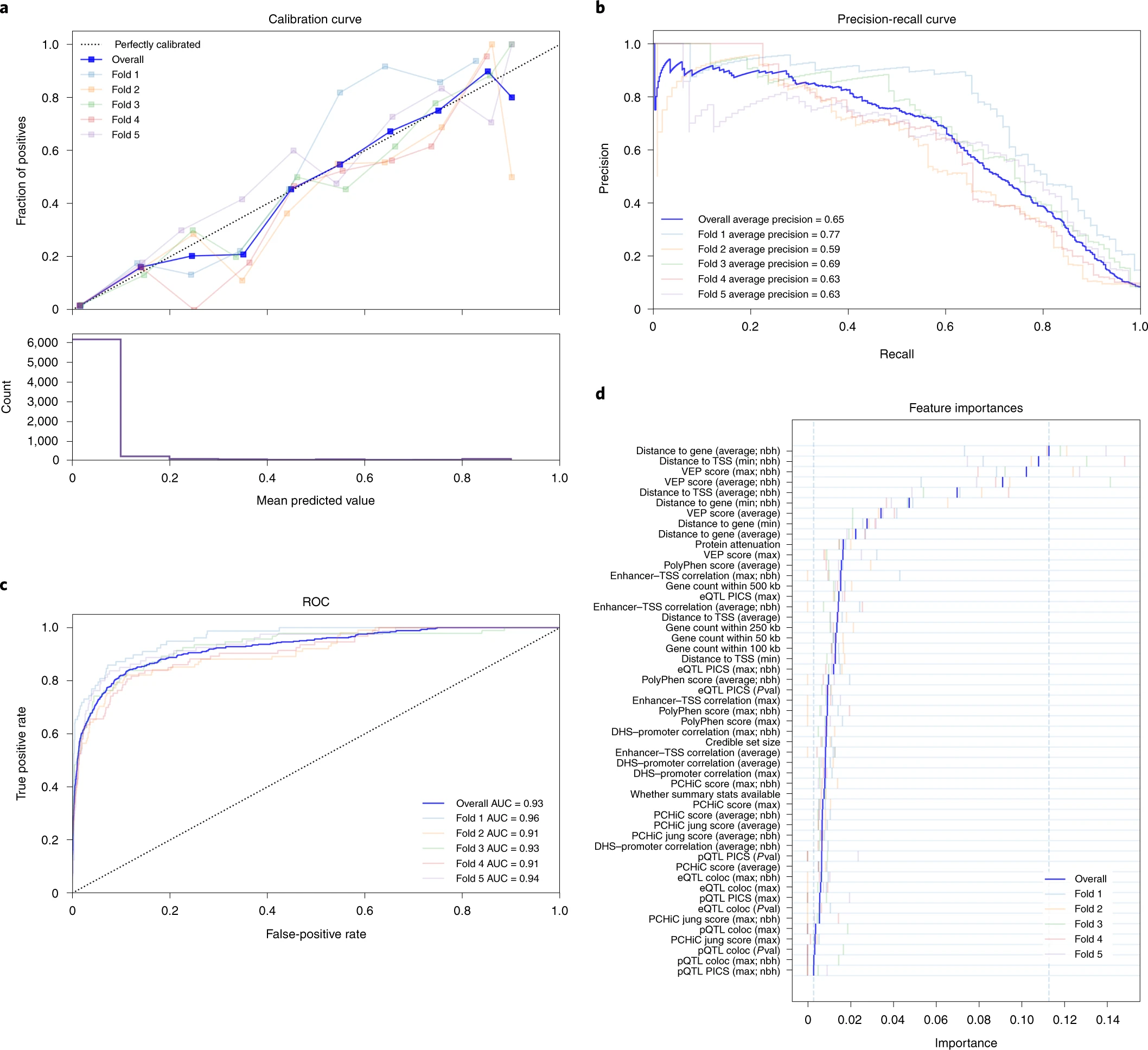
- 最后,作者将所有的结果整合在 Open Targets Genetics 网站中;
亮点:
- 开发了 L2G 模型,提高因果基因的检测性能;
- 开发了 Open Targets Genetics 网站,收录了欧洲和非欧洲的多个 GWAS 数据集以及 92 个组织/细胞类型特定 molQTL 汇总统计数据共定位结果;
文章链接:
https://www.nature.com/articles/s41588-021-00945-5
公开的资料:
- Open Targets Genetics:http://genetics.opentargets.org/
- GWAS 黄金标准基因:github.com/opentargets/genetics-gold-standards
- 代码:https://github.com/opentargets/genetics-sumstat-data; https://github.com/opentargets/genetics-finemapping;https://github.com/opentargets/genetics-colocalisation;https://github.com/opentargets/genetics-v2d-data;https://github.com/opentargets/genetics-v2g-data;https://github.com/opentargets/genetics-l2g-scoring;https://github.com/opentargets/genetics-gold-standards;https://github.com/opentargets/genetics-variant-annotation
二、精细解读文献 二
文献题目: Validation of lipid-related therapeutic targets for coronary heart disease prevention using human genetics
不想看英文题目: 使用人类遗传学验证预防冠心病的靶点
杂志和影响因子: Nat Commun (IF: 14.92; Q1)
研究意义: 全基因组生物标志物的孟德尔随机化 (MR) 已证实低密度脂蛋白胆固醇(LDL-C)增加冠心病(CHD)风险,然而高密度脂蛋白胆固醇 (HDL-C) 和甘油三酯 (TG)对冠心病的作用仍较为模糊;
结论:
- 通过全基因组生物标志物 MR 研究,在发现集中发现 LDL-C 和 TG 提高 CHD 的风险(OR CI 分别为 1.39–1.63 和 1.01–1.21),HDL 对 CHD 风险的 OR CI 为 0.90–1.01,在验证集中同样证明 LDL-C 和 TG 可提高 CHD 的风险(OR CI 分别为 1.25-1.31 和 1.14-1.32),然而 HDL 对 CHD 风险的 OR CI 为0.83–0.96,可见在单变量 MR 分析中, HDL 对 CHD 的贡献在不同的研究中是存在差异的;
- 为了评估三种脂质成分对 CHD 的独立贡献,作者在发现集中进行了多变量 MR 分析 (MVMR), 发现 LDL-C、HDL-C 和 TG 对 CHD 风险的 OR CI 分别为 (1.44-1.62、0.86-0.95、1.01–1.17);
- 随后对药物靶点进行 MR 分析,以确定对 CHD 具有影响的蛋白质。在 P < 1 × 10−6 的阈值下,发现了 341 个编码药物蛋白的基因,149 个基因与 LDL-C 相关,180 个与 HDL-C 相关,154 个与 TG 相关。在 341 个基因中,141(41%) 的基因只与一种脂质相关,101 个(30%) 的基因与两种脂质相关,100 (29%) 的基因与所有脂质相关;
- 在 341 个药物靶点中,165 个与 CHD 相关,其中 131 个药物靶点在LDL-C/TG /HDL-C 中保护作用一致;

- 药物靶点 MR 分析还发现了 40 个药物靶点被报道与脂质不良反应事件相关、另外五个基因 (PCSK9, PPARG, PPARA, NPC1L1, 和 HMGCR) 参与脂质修饰,其中 NPC1L1, HMGCR 和 PCSK9 可用于 CHD 预防;
- 为了验证 MR 结果,作者使用了 UKBB 进行验证,验证集发现了 47 个显着的 MR 结果(P值 < 0.05),其中 39/47(83%)与发现集的方向一致;
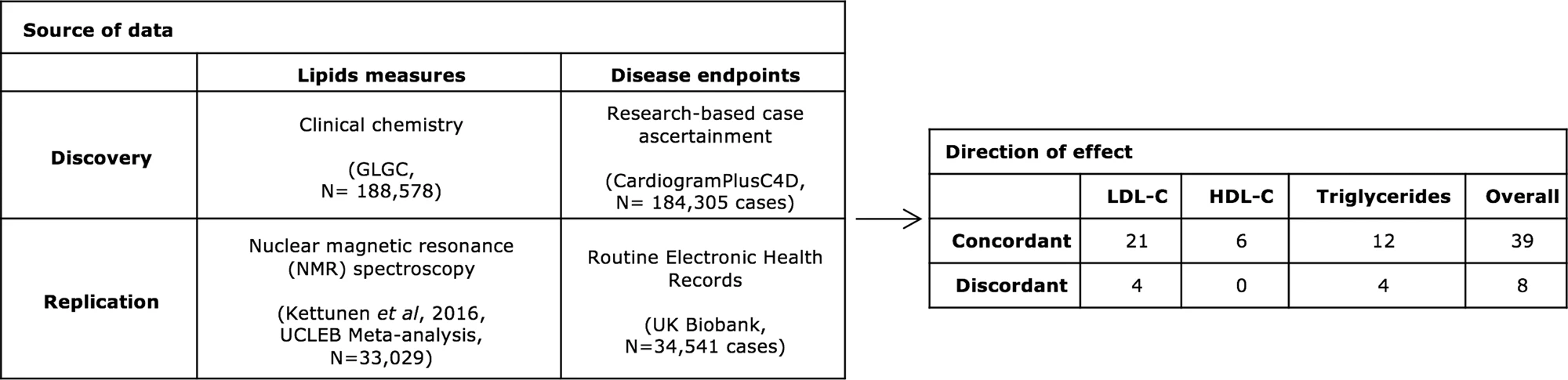
- 为了识别潜在的脂质介导途径,作者进行了多变量药物靶标 MR 分析(MVMR) ,结果发现 12 个药物靶标 (SLC12A3, APOB, APOA1, PVRL2, APOE, APOC1, CELSR2, GPR61, PCSK9, 和 CEACAM16)通过 LDL-C 影响 CHD, LPL 通过 HDL-C 影响 CHD,ALDH1A2 通过 TG 影响 CHD。另外SMARCA4 和 APOA5 通过 LDL-C 和 TG 共同影响 CHD, 而RPL7A 通过 LDL-C 和 HDL-C 共同影响 CHD;
- 对脂质和 CHD 进行共定位,发现 33 个基因座共定位,其中25 个共定位基因为药物蛋白基因;
- 为了研究药物靶基因是否在肝脏中特异性表达(肝脏为脂质代谢靶向组织),作者提取了人类蛋白质图谱的 RNAseq 数据,结果发现与其他组织相比,药物靶基因在肝脏中特异性表达;
- 为了探索药物靶基因对 CHD 潜在的其他影响,作者进行了全表型关联分析(Phenome-wide scan),发现六个药物靶基因(NDUFA13、CILP2、PVRL2、VEGFA、APOC1 和 LPL)与 2 型糖尿病相关、五个(APOC1, PVR, PVRL2, APOE 和 CEACAM16)与阿尔茨海默病相关、四个(SMARCA4、CETP、VEGFA 和 ALDH1A2)与哮喘相关,四个(APOA1、APOC3、APOA4 和 APOA5)与痛风相关;
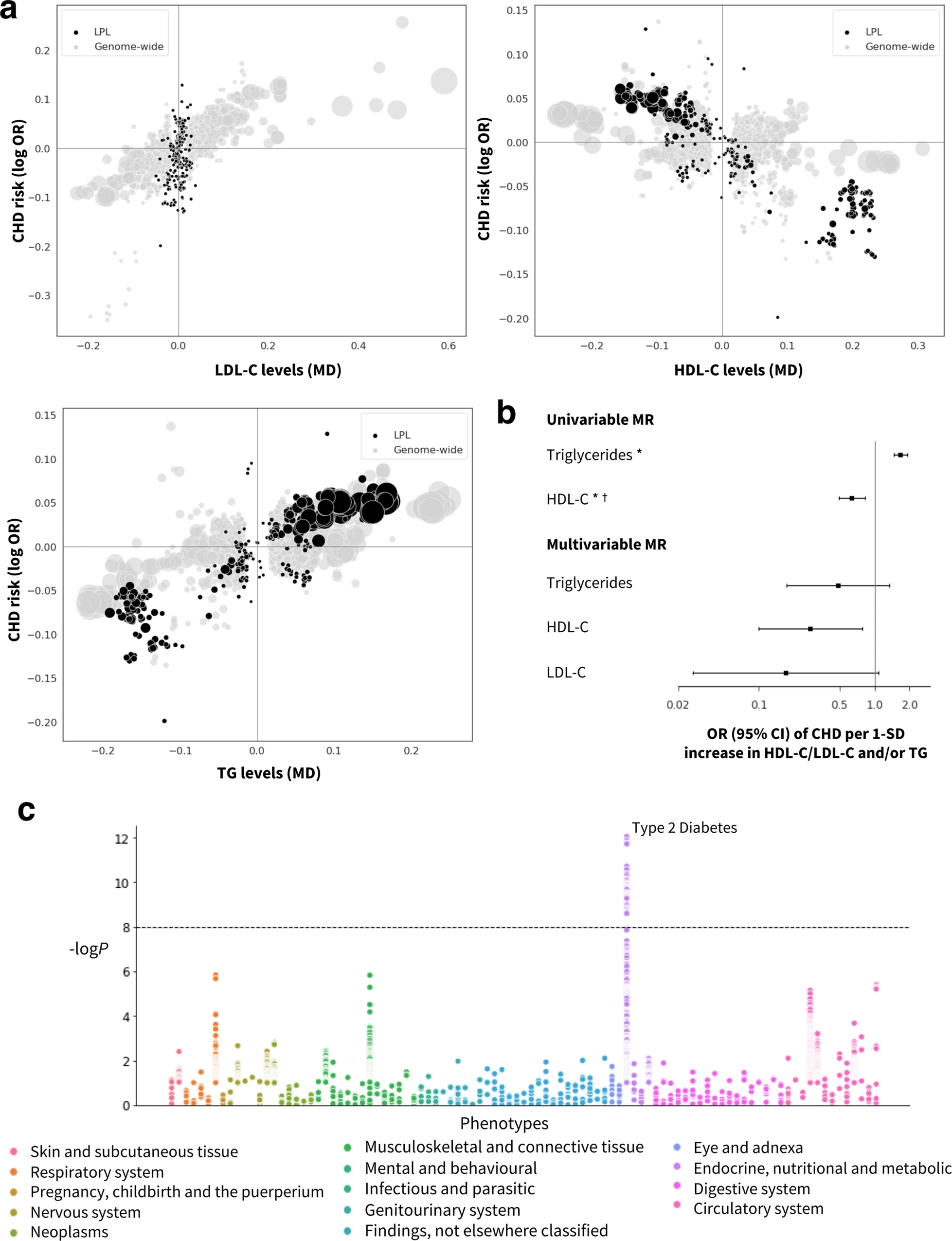
亮点: 将 GWAS 结果与药物靶标结合起来是这些年一直呼吁的方向,本文就是很好的一项例子,值得学习;
文章链接:
https://www.nature.com/articles/s41467-021-25731-z
公开的资料:
-
LDL-C, HDL-C, Triglycerides [http://lipidgenetics.org/#data-downloads-title];
-
Coronary Heart Disease [http://www.cardiogramplusc4d.org/data-downloads/];
-
Rheumatoid arthritis [https://grasp.nhlbi.nih.gov/downloads/ResultsOctober2016/Okada/]
-
Juvenile arthritis [http://ftp.ebi.ac.uk/pub/databases/gwas/summary_statistics/HinksA_23603761_GCST005528/];
-
Ankylosing spondylitis [https://ftp.ebi.ac.uk/pub/databases/gwas/summary_statistics/CortesA_23749187_GCST005529/];
-
Ulcerative colitis [http://ftp.ebi.ac.uk/pub/databases/gwas/summary_statistics/LiuJZ_26192919_GCST003045/];
-
Psoriasis [http://ftp.ebi.ac.uk/pub/databases/gwas/summary_statistics/TsoiLC_23143594_GCST005527/];
-
Crohn disease [http://ftp.ebi.ac.uk/pub/databases/gwas/summary_statistics/LiuJZ_26192919];
-
Stroke [http://www.megastroke.org/index.html];
-
Asthma [https://www.thelancet.com/journals/lanres/article/PIIS2213-2600(18)30389-8/fulltext];
-
Multiple sclerosis (https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3832895/];
-
Gout [http://ftp.ebi.ac.uk/pub/databases/gwas/summary_statistics/TinA_31578528_GCST008970/];
-
Ovarian neoplasms [http://ftp.ebi.ac.uk/pub/databases/gwas/summary_statistics/PhelanCM_28346442_GCST004462/];
-
Parkinson disease [https://drive.google.com/drive/folders/10bGj6HfAXgl-JslpI9ZJIL_JIgZyktxn];
-
Alzheimer disease (https://www.ncbi.nlm.nih.gov/pubmed/30617256];
-
Type 2 diabetes mellitus [https://www.nature.com/articles/s41588-018-0241-6];
-
Myocardial infarction [http://www.cardiogramplusc4d.org/data-downloads/];
-
Heart failure [https://www.nature.com/articles/s41467-019-13690-5];
-
Atrial fibrillation [https://www.nature.com/articles/s41588-018-0171-3];
-
Diabetic nephropathies [http://ftp.ebi.ac.uk/pub/databases/gwas/summary_statistics/vanZuydamNR_29703844_GCST005881];
-
Chronic kidney failure [https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6698888/];
-
Schizophrenia [http://www.med.unc.edu/pgc/files/resultfiles/];
-
Narcolepsy [http://ftp.ebi.ac.uk/pub/databases/gwas/summary_statistics/FaracoJ_23459209_GCST005522/];
-
Atopic dermatitis [http://ftp.ebi.ac.uk/pub/databases/gwas/summary_statistics/PaternosterL_26482879_GCST003184];
-
Biliary liver cirrhosis [http://ftp.ebi.ac.uk/pub/databases/gwas/summary_statistics/CordellHJ_26394269_GCST003129];
-
80 ICD10 main diagnoses in UK Biobank released by Neale Lab (1st August 2018, http://www.nealelab.is/uk-biobank/)
三、其他文献推荐
下面的文献也挺精彩的,但由于下不到原文,或博主时间有限,没法精细解读,故列出来供各位参阅;
当然,你们有精彩的文献想让我解读的(前提是一周内刚出炉的文献),可给我发pdf(然而可能种种原因,我不一定有时间解读,不要对我抱太高期待);
文献题目: Single-cell nuclear architecture across cell types in the mouse brain
不想看英文题目: 小鼠大脑细胞类型的单细胞核结构
杂志和影响因子: Science (IF: 41.845; Q1)
文章链接:
https://www.science.org/doi/10.1126/science.abj1966
文献题目: Exome sequencing and analysis of 454,787 UK Biobank participants
不想看英文题目: 454,787 名英国生物银行参与者的外显子组测序分析
杂志和影响因子: Nat Genet (IF: 38.33; Q1)
文章链接:
https://www.nature.com/articles/s41586-021-04103-z
文献题目: Mutational signatures in esophageal squamous cell carcinoma from eight countries with varying incidence
不想看英文题目: 八个不同发病率国家的食管鳞状细胞癌的突变特征
杂志和影响因子: Nat Genet (IF: 38.33; Q1)
文章链接:
https://www.nature.com/articles/s41588-021-00928-6
文献题目: Genome-wide association study identifies susceptibility loci for acute myeloid leukemia
不想看英文题目: 全基因组关联分析鉴定急性髓系白血病易感位点
杂志和影响因子: Nat Commun (IF: 14.92; Q1)
文章链接:
https://www.nature.com/articles/s41467-021-26551-x
文献题目: Asthma-associated genetic variants induce IL33 differential expression through an enhancer-blocking regulatory region
不想看英文题目: 哮喘相关变异位点通过增强子阻断诱导 IL33 差异表达
杂志和影响因子: Nat Commun (IF: 14.92; Q1)
文章链接:
https://www.nature.com/articles/s41467-021-26347-z
四、工具或资源类介绍
文献题目: The National Microbiome Data Collaborative Data Portal: an integrated multi-omics microbiome data resource
不想看英文题目: 集成的多组学微生物组数据库
杂志和影响因子: Nucleic Acids Res (IF: 16.97; Q1)
文章链接:
https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkab990/6414581
文献题目: BrainBase: a curated knowledgebase for brain diseases
不想看英文题目: BrainBase:脑部疾病数据库
杂志和影响因子: Nucleic Acids Res (IF: 16.97; Q1)
文章链接:
https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkab987/6414573
文献题目: TcoFBase: a comprehensive database for decoding the regulatory transcription co-factors in human and mouse
不想看英文题目: TcoFBase:人类和小鼠转录辅因子数据库
转录辅因子 (transcription co-factors, TcoFs),包括转录激酶、表观遗传蛋白和共激活因子等, 连接增强子和启动子,参与转录调控,在基因表达调控中发挥着至关重要的作用。
杂志和影响因子: Nucleic Acids Res (IF: 16.97; Q1)
文章链接:
https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkab950/6414596
文献题目: EWAS Open Platform: integrated data, knowledge and toolkit for epigenome-wide association study
不想看英文题目: EWAS 开放平台:用于全表观基因组关联分析的集成数据、知识和工具包
杂志和影响因子: Nucleic Acids Res (IF: 16.97; Q1)
文章链接:
https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkab972/6414594
文献题目: EDCNN: Identification of Genome-Wide RNA-binding Proteins Using Evolutionary Deep Convolutional Neural Network
不想看英文题目: EDCNN:使用卷积神经网络识别全基因组 RNA 结合蛋白
杂志和影响因子: Bioinformatics (IF: 5.61; Q1)
文章链接:
https://doi.org/10.1093/bioinformatics/btab739
致谢橙子牛奶糖(陈文燕),请用参考模版:We thank the blogger (orange_milk_sugar, Wenyan Chen) for XXX
感谢小可爱们多年来的陪伴, 我与你们一起成长~
