最近在做一个深度学习分类项目,想看看训练集数据的分布情况,但由于数据本身维度接近100,不能直观的可视化展示,所以就对降维可视化做了一些粗略的了解以便能在低维空间中近似展示高维数据的分布情况,以下内容不会很深入细节,但足以让你快速使用这门技术。
什么是降维可视化?
简而言之,降维是在2维或3维中展现更高维数据(具有多个特征的数据,且彼此具有相关性)的技术。
降维思想主要有两种:
- 仅保留原始数据集中最相关的变量(特征选择)。
- 寻找一组较小的新变量,其中每个变量都是输入变量的组合,包含与输入变量基本相同的信息(降维)。
什么时候需要用到降维可视化?
如果你的数据集有数十个或者数百个特征,而你想直观的看出数据集样本之间的分布情况,那么降到2维或3维来展示这种分布是一个不错的选择。
有哪些主流的降维可视化方法?
降维可视化方法其实还挺多的,但是最常见的是以下三种:
- t-SNE
t-分布式随机邻域嵌入是一种用于挖掘高维数据的非线性降维算法。 它将多维数据映射到适合于人类观察的两个或多个维度。sklearn中已有相应的实现,用起来很方便。 - PCA
主成分分析,是一种线性降维方法,虽然快,但相比非线性降维丢失的信息更多。 - LargeVis
一种在t-SNE之上提出的更快的,效果和t-SNE差不多的降维算法,项目地址:https://github.com/lferry007/LargeVis
t-SNE的原理?
我们知道,数据降维后,数据中的信息是有一定的损失量的,这个损失量在t-SNE方法中,是采用K-L散度来计算的。
K-L散度计算的是“用一个分布q来近似另一个分布p时的信息损失量”,其公式如下:

我们知道,对于一组离散型随机变量{x1,x2,...,xn},其期望值=x1* x1的概率+x2 * x2的概率+xn * xn的概率,所以上式可以用期望值表达成分布p和分布q之间的对数差值的期望,这里对数差值对应之前说的一组随机变量:

更一般的写法如下,根据log a - log b = log (a/b):

K-L散度越小,表示信息损失越小,两个分布越相近。
现在回到t-SNE,我们使用t-SNE是为了将高维数据用低维数据来表达,以便能够可视化。那么这里就涉及到2种分布,一个是高维数据的分布p,一个是低维数据的分布q,想让低维数据能够最好的表达高维的情况,就可以将K-L散度公式做为损失函数,通过最小化散度来学习出q分布下的各样本点。
目标函数:

其中:
p分布是基于高斯分布来构建的,表达了两点之间的相似度,对比高斯公式可以看到,这里用xi表示了总体均值μ,所以这里说的相似度是通过以样本点i为均值时,样本点i与其相差几个标准差来表达的:

q分布也是基于高斯分布来构建的,但是指定了标准差σ=1/√2(即我们规定用这样的分布来近似高维的分布),所以相比上面少了σ的参数项。

上面说的其实是SNE方法,t-SNE相对SNE的区别如下:
- 使用联合概率(xi和xj同时出现的概率)代替条件概率(xi出现的条件下xj出现的概率、xj出现的条件下,xi出现的概率),调整后的公式如下:

- 低维空间下,使用t分布代替高斯分布表达两点之间的相似度,调整后的q分布和梯度如下:


这样调整后,梯度计算会更加简洁,并且在这样得梯度公式下,当遇到高维空间中距离不相近,但是低维空间中距离相近的样本点,也会产生较大的梯度,让模型学习到这些点在低维空间中并不靠的近,也就是说这样会使得高维空间相似的样本在低维空间靠的更近,高维空间不相似的点在低维空间分的更开,避免SNE经常出现的拥挤(各个簇聚在一起无法区分)问题。
如何使用t-SNE?
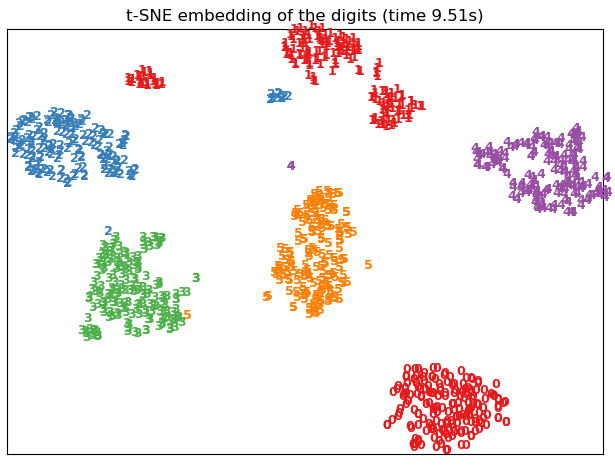
看一个对手写数字图片进行二维可视化的例子,效果如下:
代码如下:
"""
t-SNE对手写数字进行可视化
"""
from time import time
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
from sklearn.manifold import TSNE
def get_data():
digits = datasets.load_digits(n_class=6) # 取数字0-5
data = digits.data
label = digits.target
n_samples, n_features = data.shape
return data, label, n_samples, n_features
def plot_embedding(data, label, title):
x_min, x_max = np.min(data, 0), np.max(data, 0)
data = (data - x_min) / (x_max - x_min)
fig = plt.figure()
ax = plt.subplot(111)
for i in range(data.shape[0]):
plt.text(data[i, 0], data[i, 1], str(label[i]),
color=plt.cm.Set1(label[i] / 10.),
fontdict={'weight': 'bold', 'size': 9})
plt.xticks([])
plt.yticks([])
plt.title(title)
return fig
def main():
data, label, n_samples, n_features = get_data()
print('Computing t-SNE embedding')
# 降到2维
tsne = TSNE(n_components=2, init='pca', random_state=0)
t0 = time()
result = tsne.fit_transform(data)
plot_embedding(result, label,
't-SNE embedding of the digits (time %.2fs)'
% (time() - t0))
plt.show()
if __name__ == '__main__':
main()
参考资料:
http://www.datakit.cn/blog/2017/02/05/t_sne_full.html#25-不足
ok,本篇就这么多内容啦~,感谢阅读O(∩_∩)O。