1.已训练好的词向量
//自己训练词向量需要对应领域非常大的文本库,收集处理过程是很费时的,所以使用已有的资源即可。
1.1生物方面:http://bio.nlplab.org/
这里的词向量是使用word2vec工具在PubMed和PMC上文本生成的。
下载链接:http://evexdb.org/pmresources/vec-space-models/

引用论文:Distributional Semantics Resources for Biomedical Text Processing. Sampo Pyysalo, Filip Ginter, Hans Moen, Tapio Salakoski and Sophia Ananiadou. LBM 2013.
1.2化学方面:参见(6).
训练的50维的词向量,使用的数据集是以‘chemical’为搜索词,从Pubmed上下载了1918662篇MEDLINE摘要训练的,并且加上CHEMDNER语料库进行训练词向量,是用了word2vec工具,和上述是一样的。
1.3多方面https://nlp.stanford.edu/projects/glove/

a。六十亿的token,40万的字典,包括50维、100维、200维、300维的词向量。
b。等等。
其中点击Gigaword5显示:English Gigaword Fifth Edition is a comprehensive archive of newswire(新闻专线)text data that has been acquired over several years by the Linguistic Data Consortiume (LDC).
也就是说它是新闻方面的词向量。还有常见爬虫动物?以及推特数据。
2.Attending to Characters in Neural Sequence Labeling Models
这篇论文是一个独立于语料库的序列标注,并且其代码:https://github.com/marekrei/sequence-labeler
将词向量word-level和char-level结合起来,并且不是进行直接粘贴,而是通过NN训练了一个权重,减少了参数使用量。
3.Transfer learning for biomedical named entity recognition with neural networks
首先是在SSC上进行预训练,之后又转移到GSC上进行训练、验证和测试,实现了迁移学习。
代码: https://github.com/Franck-Dernoncourt/NeuroNER/
提供了进行预处理好的SSC,是Brat格式的文件:https://github.com/BaderLab/Transfer-Learning-BNER-Bioinformatics-2018/
Brat格式:标注是与被标注文档单独并且排序存储的,所以对语料库中的每一个文本文档,都有一个单独的标注文件,这两个文件通过文件命名约定关联,它们的基本名称(不带后缀的文件名)是相同的。
所有的标注都有同样的规则:每一行都包含一个标注,一个标注包括:ID、起始下标、终止下标、标签。
4.关于NeuroNER
安装过程之前博客里有过。
调用过程:
C:UsersXXXAppDataRoamingPythonPython35site-packages euroner>python __main__.py --train_model=False --use_pretrained_model=True --dataset_text_folder=./data/example_unannotated_texts --pretrained_model_folder=./trained_models/conll_2003_en //上面这个可以运行成功,看来是需要跑到那个下面去,并且直接运行main文件就可以,里面有进行构建模型的函数,就是主函数, //那么通过这个我能够知道以后也是可以这么运行的了。
5.Neural Architectures for Named Entity Recognition 2016
实现:https://github.com/glample/tagger
最早提出了把word-level和char-level的表示放在一起,并且输出层使用CRF的模型。
是通过theano实现的。
6.An attention-based BiLSTM-CRF approach to document-level chemical named entity recognition
https://github.com/lingluodlut/Att-ChemdNER
本链接中提供了,一下两个语料库:
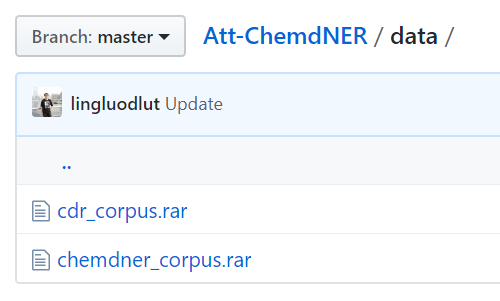
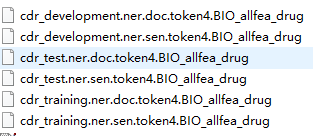
包含一下内容:
7 http://biocreative.bioinformatics.udel.edu
这里也有很多语料
8.药物不良反应
http://sideeffects.embl.de/download/
9.http://physionet.org/mimic2/
The MIMIC II (Multiparameter Intelligent Monitoring in Intensive Care) Databases contain physiologic signals and vital signs time series captured from patient monitors, and comprehensive clinical data obtained from hospital medical information systems, for tens of thousands of Intensive Care Unit (ICU) patients*.
10.Deep learning with word embeddings improves biomedical named entity recognition
https://corposaurus.github.io/corpora/
包括各种类型的生物医药数据集。
11.SSC语料库
论文:Calbc: releasing the final corpora
根据提供的语料库链接点进去就戛然而止...这个网站到底是做什么的,语料库应该不在了,现在变成一个新闻财经...。
如果真的需要的话可以给原作发邮件吧