1.代码结构
├── data // 图数据
├── inits // 初始化的一些公用函数
├── layers // GCN层的定义
├── metrics // 评测指标的计算
├── models // 模型结构定义
├── train // 训练
└── utils // 工具函数的定义
2.数据
Data: cora,Citeseer, or Pubmed,在data文件夹下:
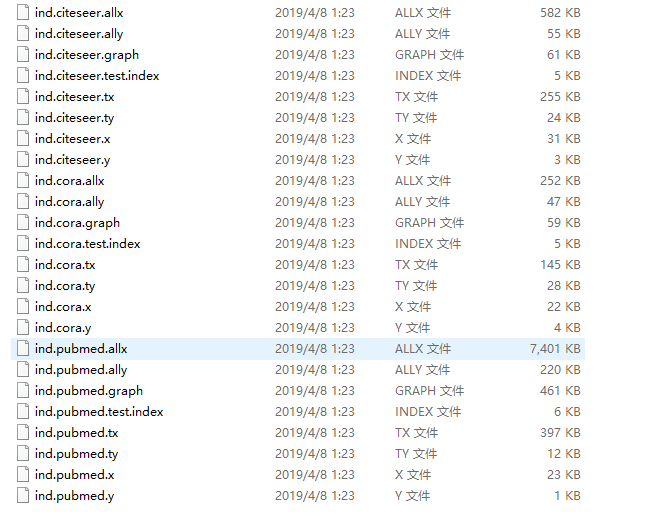
Original data:
Cora的数据集
包括2708份科学出版物,分为7类。引文网络由5429个链接组成。数据集中的每个发布都由一个0/1值的单词向量来描述,该向量表示字典中相应单词的存在或不存在。这部词典由1433个独特的单词组成。数据集中的自述文件提供了更多的细节。

CiteSeer文献分类
CiteSeer数据集包括3312种科学出版物,分为6类。引文网络由4732条链接组成。数据集中的每个发布都由一个0/1值的单词向量来描述,该向量表示字典中相应单词的存在或不存在。该词典由3703个独特的单词组成。
数据集中的自述文件提供了更多的细节。

CiteSeer for Entity Resolution
为了实体解析,CiteSeer数据集包含1504个机器学习文档,其中2892个作者引用了165个作者实体。对于这个数据集,惟一可用的属性信息是作者名。完整的姓总是给出的,在某些情况下,作者的全名和中间名是给出的,其他时候只给出首字母。
PubMed糖尿病数据库
由来自PubMed数据库的19717篇与糖尿病相关的科学出版物组成,分为三类。引文网络由44338个链接组成。数据集中的每个出版物都由一个TF/IDF加权词向量来描述,这个词向量来自一个包含500个唯一单词的字典。数据集中的自述文件提供了更多的细节。

以下以cora数据集为例:



数据集预处理:
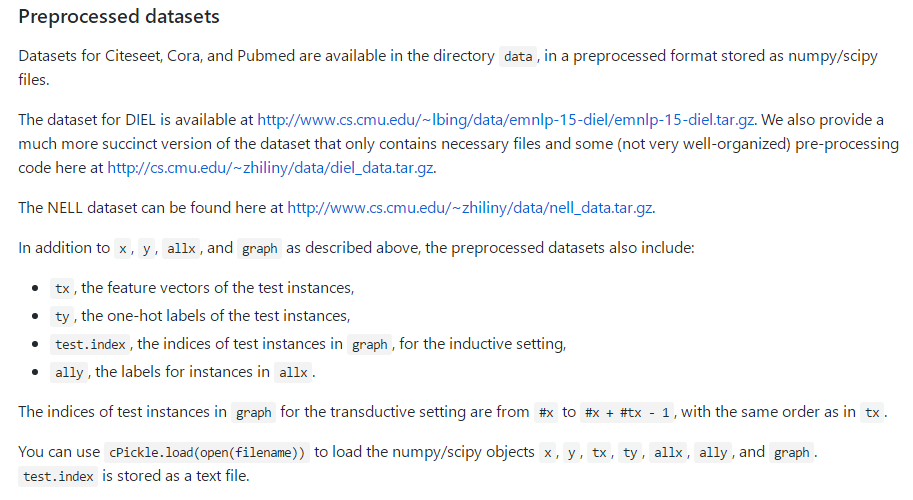
读取数据:

"""
def load_data(dataset_str):
------
Loads input data from gcn/data directory
ind.dataset_str.x => the feature vectors of the training instances as scipy.sparse.csr.csr_matrix object;
ind.dataset_str.tx => the feature vectors of the test instances as scipy.sparse.csr.csr_matrix object;
ind.dataset_str.allx => the feature vectors of both labeled and unlabeled training instances
(a superset of ind.dataset_str.x) as scipy.sparse.csr.csr_matrix object;
ind.dataset_str.y => the one-hot labels of the labeled training instances as numpy.ndarray object;
ind.dataset_str.ty => the one-hot labels of the test instances as numpy.ndarray object;
ind.dataset_str.ally => the labels for instances in ind.dataset_str.allx as numpy.ndarray object;
ind.dataset_str.graph => a dict in the format {index: [index_of_neighbor_nodes]} as collections.defaultdict
object;
ind.dataset_str.test.index => the indices of test instances in graph, for the inductive setting as list object.
All objects above must be saved using python pickle module.
:param dataset_str: Dataset name
:return: All data input files loaded (as well the training/test data).
------
names = ['x', 'y', 'tx', 'ty', 'allx', 'ally', 'graph']
objects = []
for i in range(len(names)):
with open("data/ind.{}.{}".format(dataset_str, names[i]), 'rb') as f:
if sys.version_info > (3, 0):
objects.append(pkl.load(f, encoding='latin1'))
else:
objects.append(pkl.load(f))
x, y, tx, ty, allx, ally, graph = tuple(objects)
test_idx_reorder = parse_index_file("data/ind.{}.test.index".format(dataset_str))
test_idx_range = np.sort(test_idx_reorder)
if dataset_str == 'citeseer':
# Fix citeseer dataset (there are some isolated nodes in the graph)
# Find isolated nodes, add them as zero-vecs into the right position
test_idx_range_full = range(min(test_idx_reorder), max(test_idx_reorder)+1)
tx_extended = sp.lil_matrix((len(test_idx_range_full), x.shape[1]))
tx_extended[test_idx_range-min(test_idx_range), :] = tx
tx = tx_extended
ty_extended = np.zeros((len(test_idx_range_full), y.shape[1]))
ty_extended[test_idx_range-min(test_idx_range), :] = ty
ty = ty_extended
features = sp.vstack((allx, tx)).tolil()
features[test_idx_reorder, :] = features[test_idx_range, :]
adj = nx.adjacency_matrix(nx.from_dict_of_lists(graph))
labels = np.vstack((ally, ty))
labels[test_idx_reorder, :] = labels[test_idx_range, :]
idx_test = test_idx_range.tolist()
idx_train = range(len(y))
idx_val = range(len(y), len(y)+500)
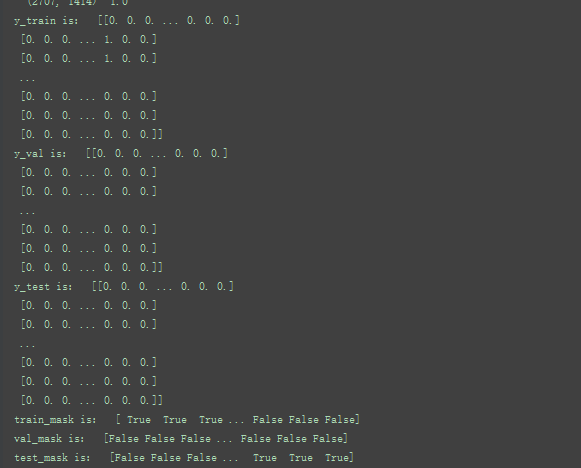
train_mask = sample_mask(idx_train, labels.shape[0])
val_mask = sample_mask(idx_val, labels.shape[0])
test_mask = sample_mask(idx_test, labels.shape[0])
y_train = np.zeros(labels.shape)
y_val = np.zeros(labels.shape)
y_test = np.zeros(labels.shape)
y_train[train_mask, :] = labels[train_mask, :]
y_val[val_mask, :] = labels[val_mask, :]
y_test[test_mask, :] = labels[test_mask, :]
return adj, features, y_train, y_val, y_test, train_mask, val_mask, test_mask
"""
知识点1:
那么为什么需要序列化和反序列化这一操作呢? 便于存储。序列化过程将文本信息转变为二进制数据流。这样就信息就容易存储在硬盘之中,当需要读取文件的时候,从硬盘中读取数据,然后再将其反序列化便可以得到原始的数据。在Python程序运行中得到了一些字符串、列表、字典等数据,想要长久的保存下来,方便以后使用,而不是简单的放入内存中关机断电就丢失数据。python模块大全中的Pickle模块就派上用场了,它可以将对象转换为一种可以传输或存储的格式。 loads()函数执行和load() 函数一样的反序列化。取代接受一个流对象并去文件读取序列化后的数据,它接受包含序列化后的数据的str对象, 直接返回的对象。
import cPickle as pickle
pickle.dump(obj,f) # 序列化方法pickle.dump()
pickle.dumps(obj,f) #pickle.dump(obj, file, protocol=None,*,fix_imports=True) 该方法实现的是将序列化后的对象obj以二进制形式写入文件file中,进行保存。它的功能等同于 Pickler(file, protocol).dump(obj)。
pickle.load(f) #反序列化操作: pickle.load(file, *,fix_imports=True, encoding=”ASCII”. errors=”strict”)
pickle.loads(f)
回顾以下:
cora数据集:包括2708份科学出版物,分为7类。引文网络由5429个链接组成。数据集中的每个发布都由一个0/1值的单词向量来描述,该向量表示字典中相应单词的存在或不存在。这部词典由1433个独特的单词组成。
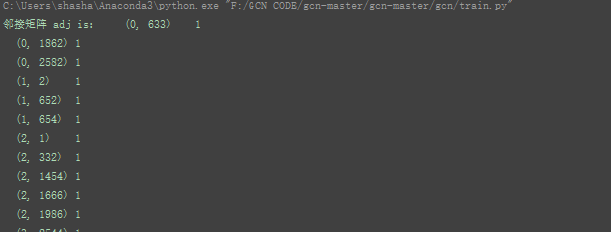
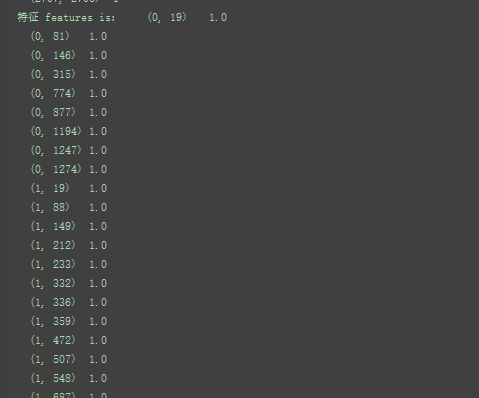
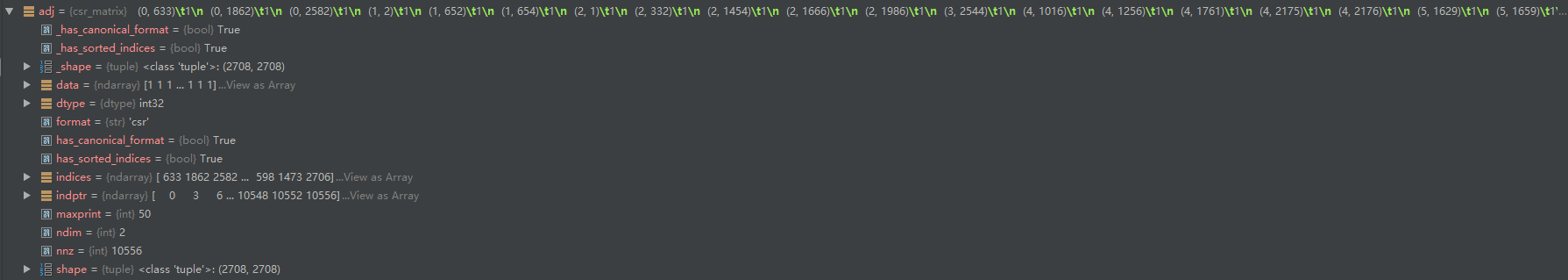
邻接矩阵adj:27082708 对应2708份科学出版物,以及对应的连接
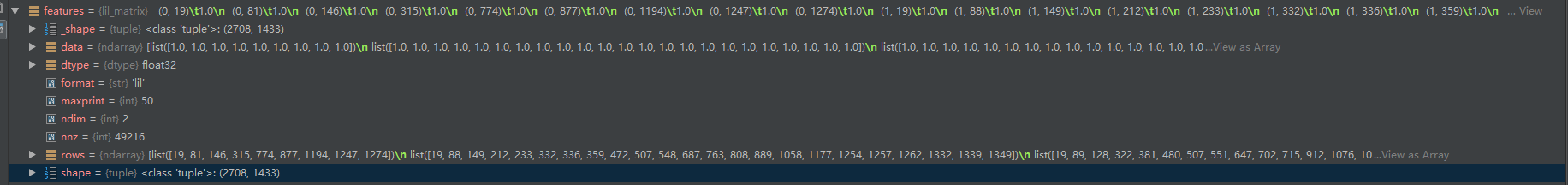
特征feature :27081433 ,这部词典由1433个独特的单词组成。
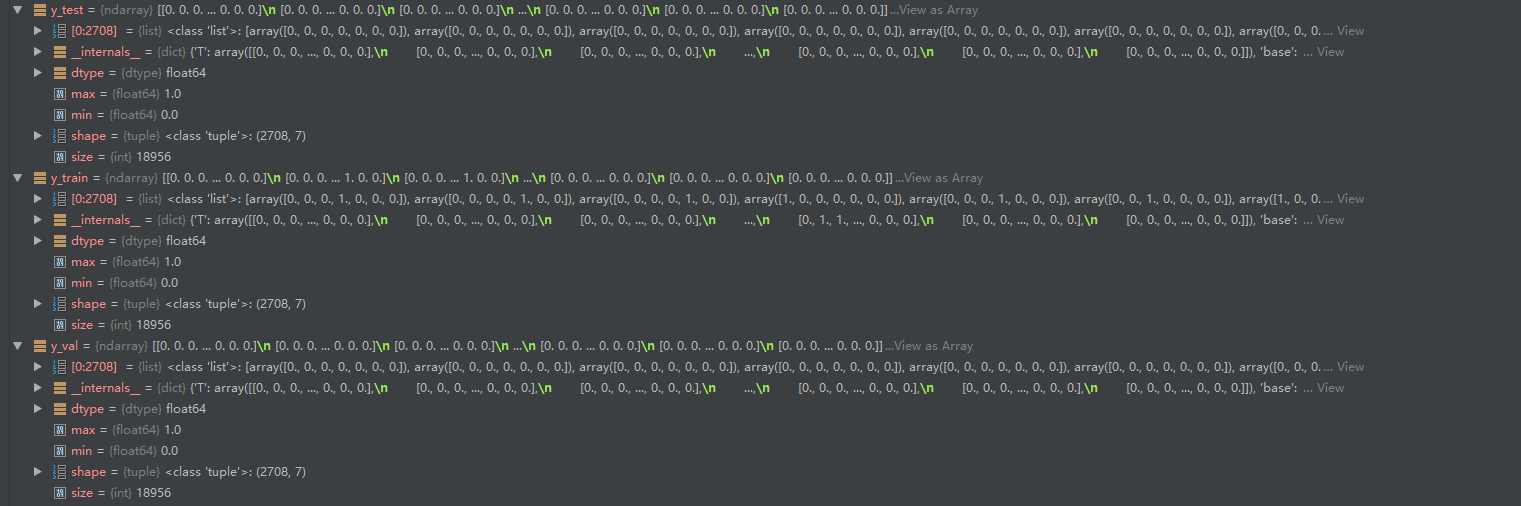
7对应7类
接下来做一些处理:
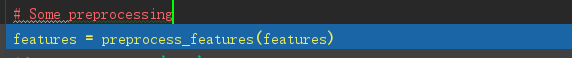
行规格化特征矩阵并转换为元组表示:

处理特征:将特征进行归一化并返回tuple (coords, values, shape)
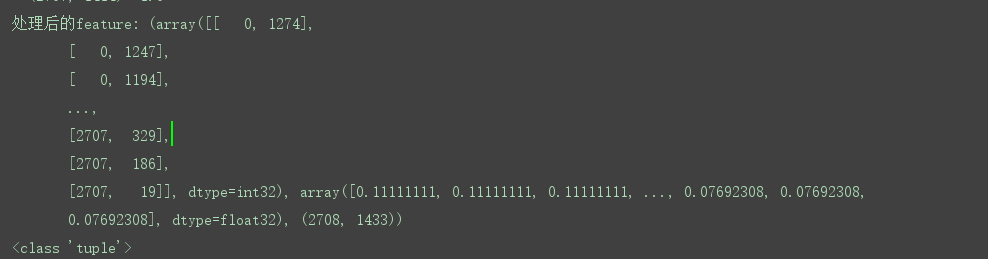
模型的选择
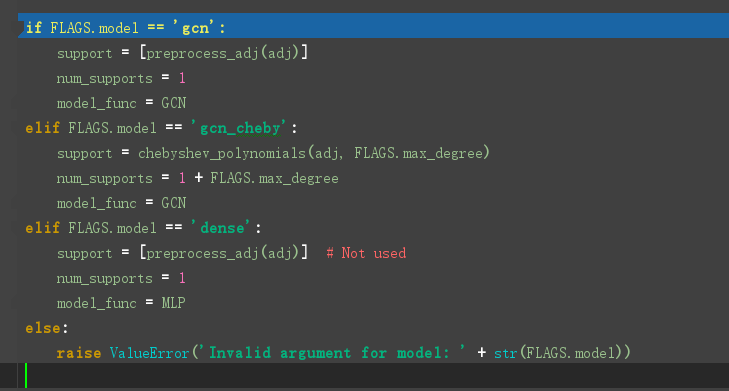

GCN定义在model.py文件中,model.py 定义了一个model基类,以及两个继承自model类的MLP、GCN类。
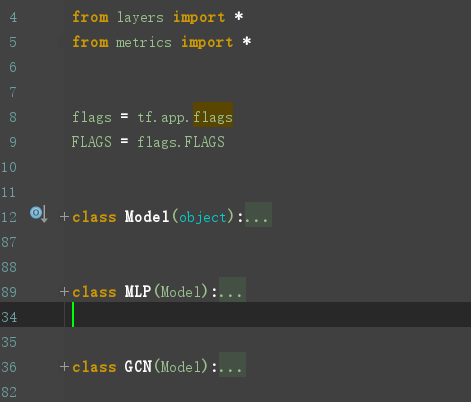
重点来看看GCN类的定义:
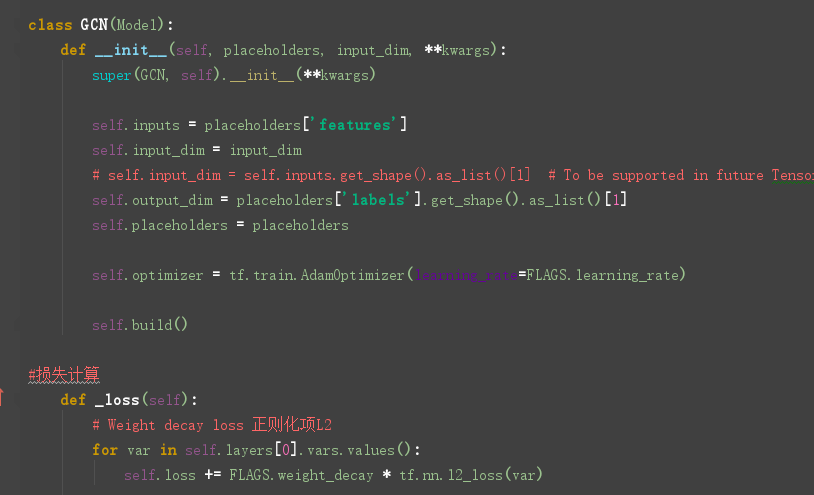

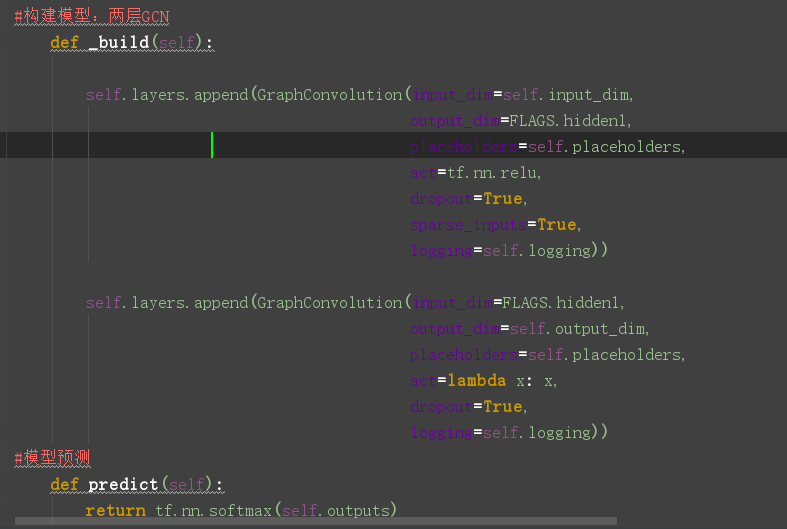
看一下图卷积层的定义:在layer.py文件中,
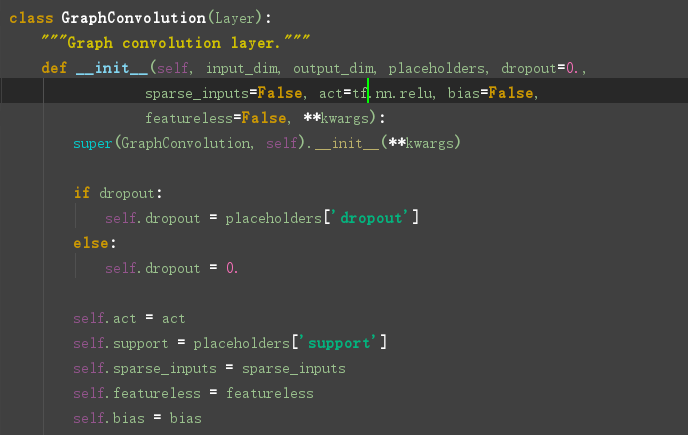

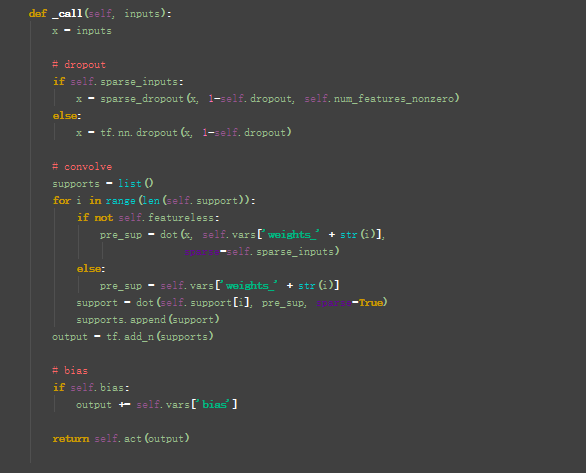
接下来:
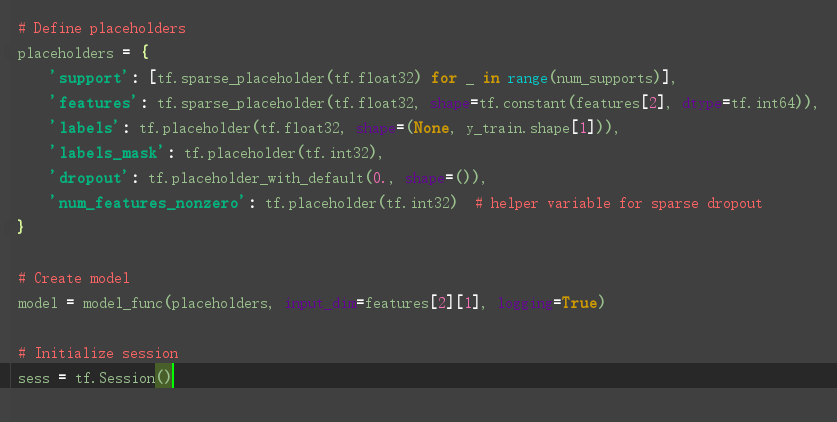

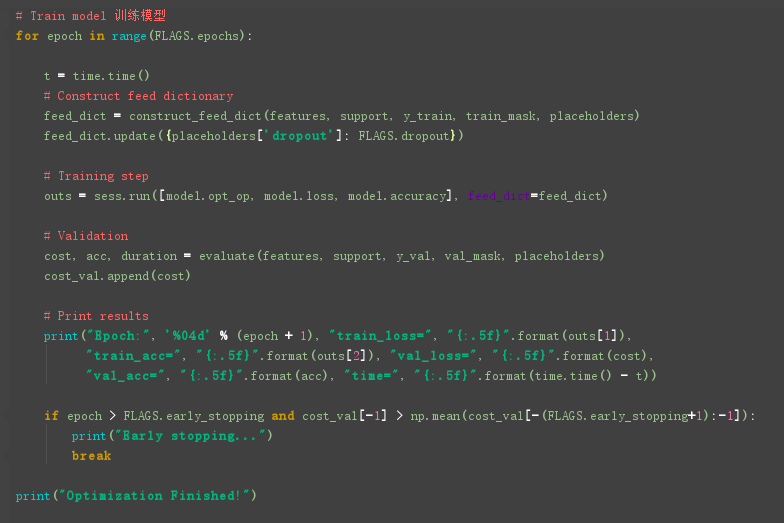
然后训练模型:
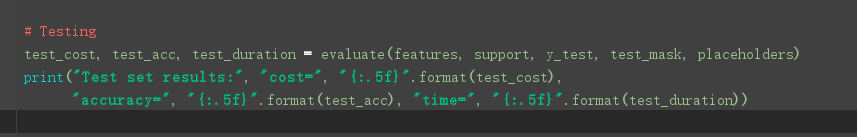
细节补充1:
参考:https://blog.csdn.net/weixin_36474809/article/details/89379727